Serpinas: trampas moleculares para capturar proteasas
La familia de las serpinas comprende un conjunto de proteínas estructuralmente similares, pero con funciones diversas. Nombrados originalmente por su función como inhibidores de serina proteasas (su nombre viene del inglés serin protease inhibitors)
están involucradas en el almacenamiento y transporte de proteínas y otras funciones. Se han identificado 37 serpinas en el cuerpo humano, de las cuales 30 son inhibidoras de proteasas funcionales.
Por otra parte, las serpinas participan en la inflamación, la función inmune, la tumorigénesis, la coagulación de la sangre, la demencia y la metástasis del cáncer (Figura 1). En los últimos años, el estudio de estas proteínas
se ha centrado en la búsqueda de aplicaciones como biomarcadores potenciales y objetivos terapéuticos.
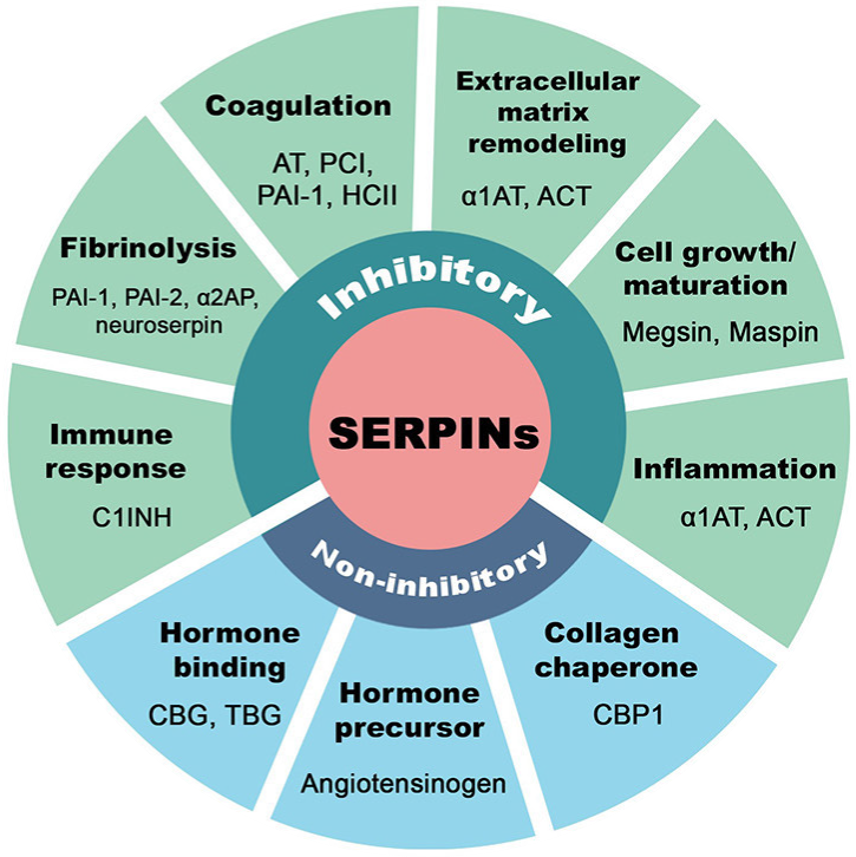 Figura 1. Funciones reguladoras de las serpinas. Fuente: Sanrattana et al., 2019.
Figura 1. Funciones reguladoras de las serpinas. Fuente: Sanrattana et al., 2019.
Estructura: secuencias y dominios.
Las serpinas son una familia de inhibidores de proteasas de alto peso molecular constituidas por 350–400 residuos de aminoácidos. Su peso molecular varía entre 40 y 100 kDa debido a las diferencias en su perfil de glicosilación. Tienen
un alto nivel de expresión en el hígado, aunque también se manifiestan de manera ubicua en todo el cuerpo.
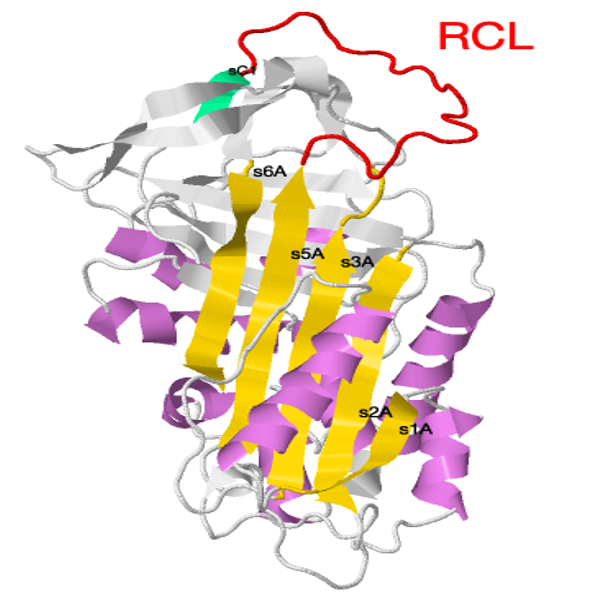
Las serpinas se pliegan en forma de un único dominio constituido por
7-9 hélices α (en violeta en el siguiente modelo) y 3 láminas β (A, B y C). Estas se distribuyen mayoritariamente en un lado de la lámina-β A, que está compuesta por cinco hebras A
(s1A, s2A, s3A, s5A y s6A) (en dorado en el modelo) las cuales se colocan en el centro de la molécula. Algunas serpinas sufren extensiones a nivel de sus extremos, lo cual incrementa considerablemente su tamaño.
Estructura de las serpinas.
En la parte superior de la molécula expone un bucle flexible, también conocido como bucle del centro reactivo (RCL de sus siglas en inglés) (en rojo). Este se caracteriza por su longitud
(entre 20 y 24 residuos) y flexibilidad, de modo que trata de simular la estructura del sustrato. Una vez unida a la proteasa, establece una conformación rígida. Esto favorece el acoplamiento a las serín-proteasas,
uniéndose fuertemente y de forma reversible a estas proteínas y evitando de esta forma que se produzcan cambios conformacionales en la proteína diana una vez se ha producido el ataque proteolítico.
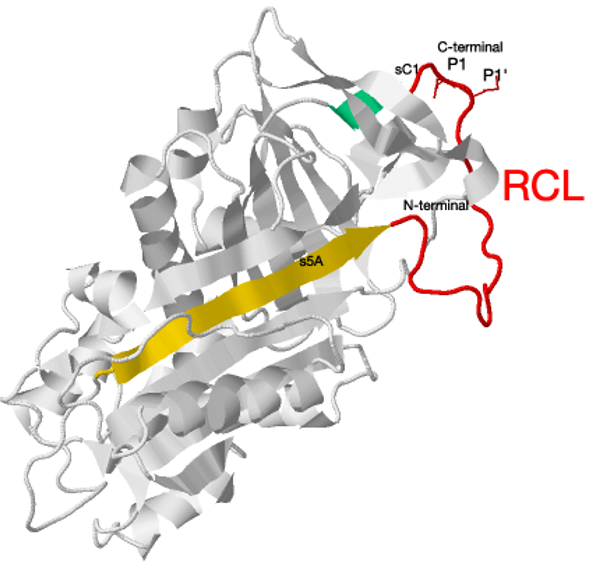
En el siguiente enlace se muestra cómo el RCL está unido a la hebra s5A (en dorado) en el extremo N-terminal y a la sC1 su el
extremo C-terminal. Además, contiene un sitio de escisión enzimática (P1-P1´) que se encuentra cerca del extremo C-terminal del RCL, según se muestra en el siguiente modelo.
Bucle del centro reactivo (RCL) de la serpina α1-antitripsina mostrando los residuos P1 y P1´.
La especificidad de las serpinas no solo está determinada por sus secuencias RCL, sino también por los exositios que son sitios de unión secundarios alejados del sitio de escisión RCL.
Conformación y cambios conformacionales
La conformación de las serpinas descrita en los modelos anteriores es una conformación metaestable, con el RCL móvil. En presencia de su sustrato, las serpinas tienen la capacidad de establecer una conformación hiperestable sin la ayuda de chaperonas.
Inhibidores de las serpinas regulan las actividades de las proteasas circulantes. Las serpinas inhiben a las serín proteasas mediante la acilación de la serina de su centro activo. Antes de que se produzca la desacetilación y
la proteólisis completa de la serpina, se desencadenan cambios de conformación masivos en la serpina mientras se mantiene el vínculo covalente entre la proteasa y la serpina. Al ejecutar su función, las serpinas actúan como “ratoneras”
moleculares, donde el RCL es el “cebo” y las proteasas diana son “ratones”. El proceso de inhibición empieza cuando una proteasa reconoce el cebo y se une a la serpina, formando un complejo reversible Michaelis-Menten,
mostrado en el siguiente modelo.
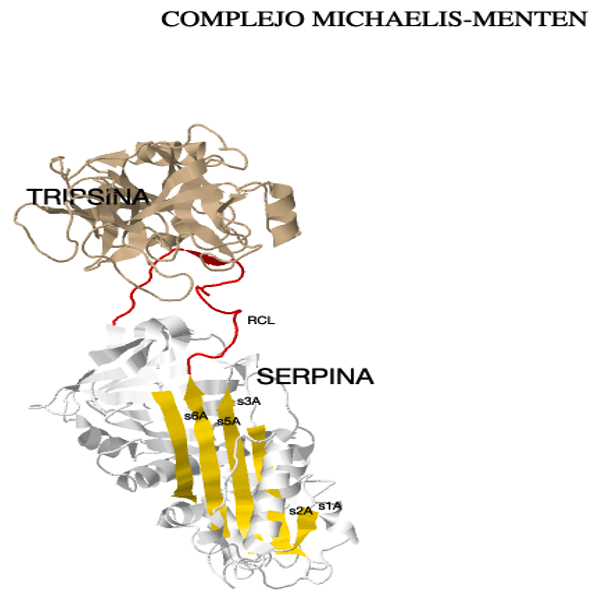
Complejo Michaelis-Menten de la serpina.
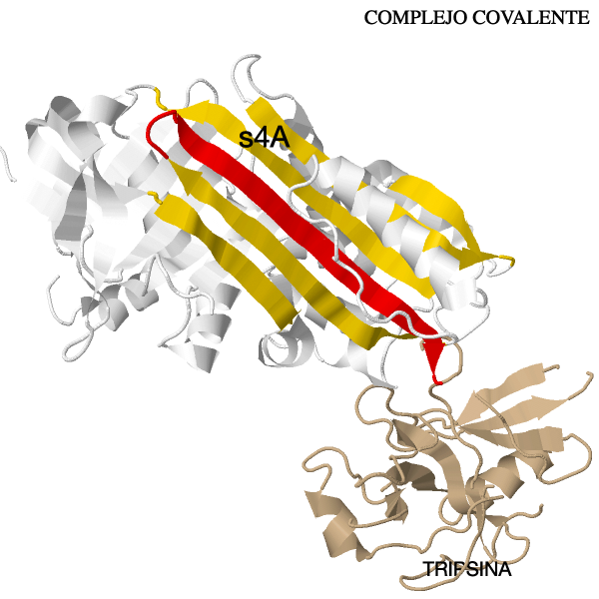
Después, la serpina sufre importantes cambios conformacionales siguiendo unida a la proteasa: el RCL se dispone en el centro de la molécula como la cuarta hebra central (s4A , en rojo en el modelo inferior) de la lámina-β A y desplaza a la hebra 1 para conseguirlo. Este cambio conformacional tiene lugar de forma espontánea, adquiriendo una conformación “latente” más estable que la nativa y que
presenta una vida media alta. En el siguiente modelo se observa la conformación hiperestable de la serpina α1-antitripsina unida a su sustrato tripsina. Esta conformación hiperestable tiene una energía libre muy negativa
debido a que el RCL es capaz de insertarse en la lámina sin necesidad de desplazar a la hebra sC1. En este estado, las serpinas aprovechan la diferencia que hay entre la energía libre de su estado activo y
la del complejo serina-proteasa, para atacar a la proteasa y producir una distorsión en el sitio activo.
Conformación hiperestable de la serpina (complejo covalente).
Cuando la proteasa anclada escinde el enlace entre los residuos P1 y P1´de la serpina, el bucle C-terminal del RCL se inserta en el cuerpo de la serpina, entre sus hojas-β A. Esto conduce, como se ha visto
más arriba, a la formación de la
s4A y a una hoja-β A antiparalela completa. Si la inserción del bucle es lo suficientemente rápida, el sitio activo de la enzima se distorsiona e inactiva, dejando el complejo enzima-serpina unido covalentemente (ver modelo
anterior y Figura 2, estructura D ). En cambio, si es demasiado lenta, el enlace covalente se rompe antes de que el sitio activo de la enzima se desactive y la serpina se consume como sustrato (Figura 2, estructura E ).
La relación entre ambas posibilidades se expresa como la estequiometría de la inhibición y debería ser próxima a 1 para que las serpinas sean inhibidores potentes.
 Figura 2. Resumen de los cambios conformacionales que sufre la serpina. Fuente: Sanrattana et al., 2019.
Figura 2. Resumen de los cambios conformacionales que sufre la serpina. Fuente: Sanrattana et al., 2019.
Para finalizar esta parte, se pude visualizar un vídeo de Youtube de “The International Society on Thrombosis and Haemostasis” que ilustra desde el minuto 6:25 hasta el 9:26 el mecanismo de acción resumido anteriormente:
Mecanismo de la serpina
(vídeo)
Ventajas y desventajas del mecanismo "trampa de ratón" de las serpinas
Este mecanismo funciona como una trampa molecular capaz de eliminar la función enzimática de la proteasa. No obstante, el hecho de que estos inhibidores presenten un mecanismo tan sofisticado de plegamiento presenta ciertas desventajas,
pues determinadas mutaciones inducen a la acumulación de intermediarios y la formación de polímeros no funcionales. En diferentes estudios se ha demostrado que la modificación de la longitud del RCL en uno o dos residuos afecta drásticamente
a la estabilidad y la eficacia de inhibición.
Las ventajas del mecanismo de la serpina sobre el mecanismo estándar de llave-cerradura son múltiples e incluyen: la capacidad de regular la actividad alterando la conformación o accesibilidad del RCL, el complejo covalente es
irreversible, el cambio en la estructura de serpinas y proteasas deja disponibles complejos de cofactores y sustratos.
• Heparina: conformación de las serpinas
La actividad de varias serpinas está modulada por glicosaminoglicanos como la heparina. En general, la heparina sirve para unir la serpina con la proteasa, mejorando así la tasa de formación de complejos. La mayor parte de la
mejora de la tasa en estos casos es proporcionada por el cambio conformacional en la serpina (alosterio). Para que el alosterio contribuya a la aceleración de la heparina de la función serpina, el estado nativo de la serpina debe estar
en una conformación de baja actividad, y la nueva conformación unida a heparina debe permitir de alguna manera la formación de complejos de reconocimiento productivo (Michaelis) con proteasas objetivo.
Mutaciones de las serpinas
Las serpinas son altamente susceptibles a mutaciones que perturban su función. Los ejemplos clásicos son mutaciones en el RCL que cambian la especificidad, como la variante de Pittsburgh de α1antitripsina (α1AT)
que lo convierte en un inhibidor de la trombina, o la eliminación de la función inhibitoria al ralentizar la incorporación de RCL en la lámina β A, como la mutación Cambridge II en α1AT. Otra causa común
de deficiencia de serpinas es la disminución/ausencia de secreción de la proteína activa debido a mutaciones que afectan la capacidad de las serpinas para plegarse en el estado nativo metaestable.
Estas mutaciones pueden ocurrir en casi cualquier parte de la serpina y normalmente están asociadas con la formación y acumulación de polímeros estables dentro del retículo endoplásmico de las células secretoras. La polimerización
siempre conduce a niveles más bajos de serpina secretada, y en los casos en que la concentración de las serpinas es crítica, la deficiencia heterocigótica conduce a la enfermedad, como la trombosis para 1AT, y solo los portadores
homocigotos de mutaciones desarrollan enfermedad de pérdida de función. Ocasionalmente, la acumulación de polímeros de serpinas es tóxica para las células y provoca daño tisular a través de un mecanismo desconocido de ganancia de función.
Aunque la incidencia de la enfermedad causada por la polimerización de serpinas es baja, ha habido un gran interés en el fenómeno, debido en parte a su clasificación como una enfermedad conformacional, junto
con las enfermedades de Alzheimer, Huntington y Parkinson, y las encefalopatías priónicas.
Por último, cuando se producen procesos de proteolisis se genera el péptido C-36, que en solución presenta una configuración desestructurada. Tras el procesamiento proteolítico, se generan fibrillas amiloides altamente ordenadas y estructuralmente similares. Este fenómeno se ve favorecido en caso de adicionar heparina, que potencia su conversión en agregados de proteína beta amiloides, los cuales se relacionan con patologías como las nombrados anteriormente.
Guion elaborado por Celia Mena Santano, Mario A. Monzón Naranjo, Yasmina Rocío Luzardo, Nerea Vidaña Bedera, alumnos de Química e Ingeniería de Proteínas, 3er curso del Grado en Biotecnología, curso 2019/20.
Cuestiones
Responder al cuestionario correspondiente en la página de Studium
Referencias
Elliott, P.R., Lomas, D.A., Carrell, R.W. & Abrahams, J.P. (1996) Inhibitory conformation of the reactive loop of alpha 1-antitrypsin. Nat.Struct.Mol.Biol. 3:676-681
Gettins, P.G.W. & Olson, S.T. (2016). Inhibitory serpins. New insights into their folding, polymerization, regulation and clearance. Biochemical Journal. 473:2273-2293. doi: 10.1042/BCJ20160014
Goodsell D., RCSB PDB Molecule of the Month. doi: 10.2210 / rcsb_pdb / mom_2004_5
Heit, C., Jackson, B.C., McAndrews, M., Wright, M.W., Thompson, D.C., Silverman, G.A., et al. (2013) Update of the human and mouse SERPIN gene superfamily. Hum Genomics 7, 22. doi: 10.1186/1479-7364-7-22
Huntington, J.A. (2011) Serpin structure, function and dysfunction. Journal of Thrombosis and Haemostasis 9:26-34. doi:10.1111/j.1538-7836.2011.04360.x
PDB www.rcsb.org, H.M. Berman, J. Westbrook, Z. Feng, G. Gilliland, T.N. Bhat, H. Weissig, I.N. Shindyalov, P.E. Bourne (2000) The Protein Data Bank URL: Nucleic Acids Research, 28: 235-242.
Risør, M.W., Juhl, D.W., Bjerring, M., Mathiesen, J., Enghild, J. J., Nielsen, N.C., & Otzen, D.E. (2017). Critical Influence of Cosolutes and Surfaces on the Assembly of Serpin-Derived Amyloid Fibrils. Biophysical journal, 113(3),
580–596. doi:10.1016/j.bpj.2017.06.030
Sanrattana, W., Maas, C. & de Maat, S. (2019). SERPINs-From trap to treatment. Frontiers in Medicine, 6:25. doi:10.3389/fmed.2019.00025S