INTRODUCCIÓN Y FUNCIÓN
Las lipoproteínas son complejos macromoleculares de proteínas transportadoras específicas, denominadas apolipoproteínas, con diversas combinaciones de fosfolípidos, colesterol, ésteres de colesterol y triacilgliceroles. Su función
es transportar moléculas lipídicas de naturaleza hidrofóbica de unos órganos a otros en el medio acuoso del plasma sanguíneo. Se clasifican en función de su densidad en quilomicrones, VLDL, IDL, LDL y HDL (Tabla 1 y Figura 1). Cada
una de ellas tiene una función específica, determinada por su lugar de síntesis, composición lipídica y contenido en apolipoproteínas.
Tabla 1. Clases principales de lipoproteínas plasmáticas humanas: algunas propiedades
Composición (% peso seco)
Lipoproteína
Densidad(g/mL)
Proteínas
Fosfolípidos
Colesterol libre
Ésteres de colesterol
Triacilgliceroles
Apoproteínas
Quilomicrones
<1,006
2,5
9
3
5
89
C-II, C-III, E
VLDL
0,95-1,006
10
20
10
15
65
B-100, C-I, C-II, C-III, E
IDL
1,006-1,019
20
22
8
30
22
B-100, C-I, C-II, C-III, E
LDL
1,019-1,063
25
20
10
40
10
B-100
HDL
1,063-1,210
55
35
4
12
4
A-I, A-II, C-I,C-II, C-III, E
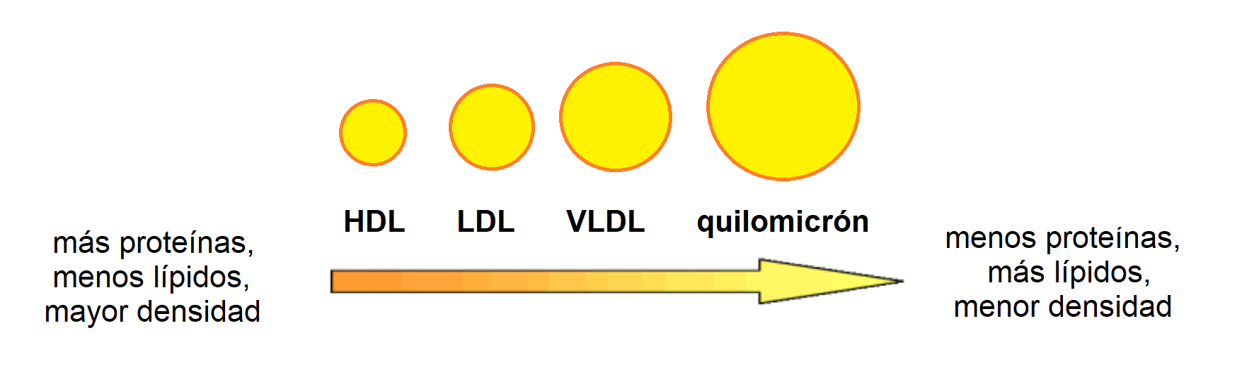 Figura 1. Características de las lipoproteínas. Tomado de la página Biomodel.
Figura 1. Características de las lipoproteínas. Tomado de la página Biomodel.
HDL
Las HDL se encargan del transporte inverso del colesterol, es decir, extraen el exceso de colesterol de los tejidos y lo transportan al hígado para que se excrete en la bilis (Figura 2).
Las HDL se producen en hígado e intestino como pequeñas partículas discoidales ricas en proteínas y se van constituyendo en el plasma a partir de componentes de la degradación de otras lipoproteínas.
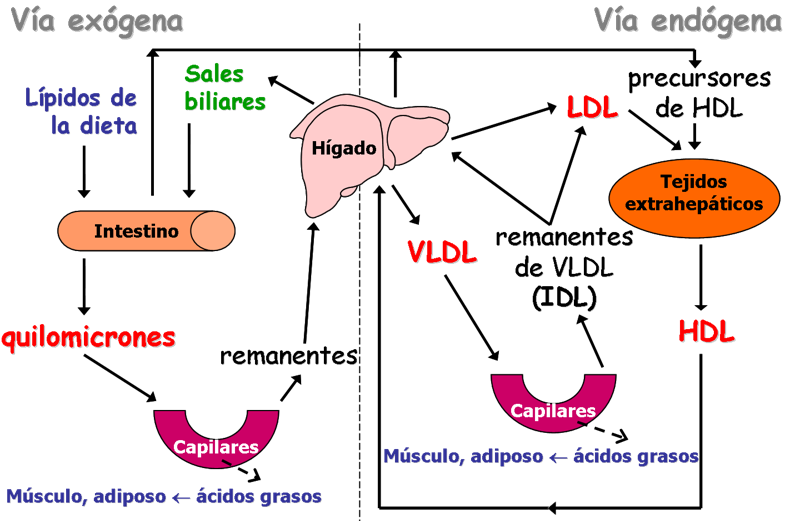 Figura 2. Función de las lipoproteínas. Tomado de la página Biomodel.
Figura 2. Función de las lipoproteínas. Tomado de la página Biomodel.
Las HDL extraen el colesterol de las membranas celulares y lo convierten en ésteres de colesterol por acción de su LCAT (Lecitin Colesterol Acil Transferasa) asociada que es activada por la ApoA-I. El trasvase de colesterol y fosfolípdios
de las células a estas lipoproteínas pobres en lípidos está mediado por un transportador de membrana denominado ABCA1 (“ATP-binding cassette transporter”).
La degradación de las HDL tiene lugar en el hígado tras su unión a una proteína de membrana, la SR-BI (“scavenger receptor class B type I”). Los ésteres de colesterol de las HDL se transfieren al hepatocito y la lipasa hepática
hidroliza los triacilgliceroles de estas lipoproteínas, mientras que la ApoA-I se recicla para formar nuevas HDL.
A continuación, se describen los aspectos estructurales más relevantes de tres proteínas importantes en el metabolismo de las HDL, la ApoA-I, ABCA1 y SR-BI.
APOA-I
La ApoA-I es una proteína de 28 kDa que contiene 243 residuos. Está codificada por los exones 3 y 4 del gen Apolipoproteína en el cromosoma 11. Forma parte de lipoproteínas como los quilomicrones (ver tabla 1) y es la principal apoproteína de las HDL,
tanto circulantes como nacientes. Supone el 70% del contenido proteico de las HDL y determina la estructura y función de estas lipoproteínas:
● Promueve el flujo de colesterol desde las membranas de las células periféricas al hígado.
● Activa la LCAT.
● Es responsable del reconocimiento de la HDL por los receptores de
HDL del hígado.
La ApoA-I debe ser flexible y así poder acomodarse a los cambios de tamaño que sufren las HDL.
HDL NACIENTES
Las HDL nacientes presentan 160 moléculas de dioleoilfosfatidilcolina (DOPC) y 2 cadenas de ApoA-I. Existen diferentes modelos propuestos para explicar su diversidad estructural que se resumen en las figura 3 A, B, C, D y E,
representándose en cada uno de los modelos la
cadena A en rojo, la
cadena B en azul, y en verde los
lípidos:
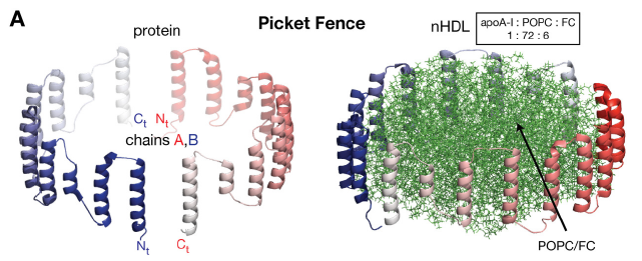 Figura 3A. Modelo valla de estacas (Picket Fence): en este modelo las alfa-hélices se disponen de manera similar a las alfa-hélice transmembrana. Este modelo ha sido posteriormente refinado.
Figura 3A. Modelo valla de estacas (Picket Fence): en este modelo las alfa-hélices se disponen de manera similar a las alfa-hélice transmembrana. Este modelo ha sido posteriormente refinado.
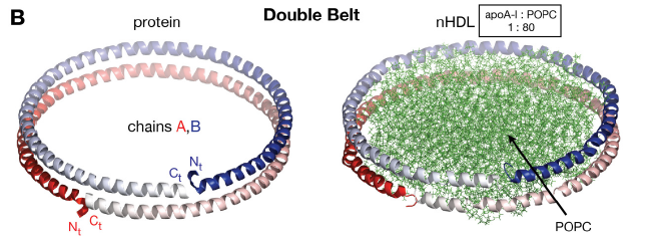 Figura 3B. Modelo cinturón doble (Double belt): este modelo cobró fuerza tras la obtención cristalográfica de la ApoA-I libre de lípidos, truncada en el extremo N-terminal (ver el modelo más abajo). Se trata de un dímero de ApoA-I
en el que se encuentra una cadena sobre otra de manera antiparalela. Es el modelo más aceptado, pero se han propuesto modificaciones.
Figura 3B. Modelo cinturón doble (Double belt): este modelo cobró fuerza tras la obtención cristalográfica de la ApoA-I libre de lípidos, truncada en el extremo N-terminal (ver el modelo más abajo). Se trata de un dímero de ApoA-I
en el que se encuentra una cadena sobre otra de manera antiparalela. Es el modelo más aceptado, pero se han propuesto modificaciones.
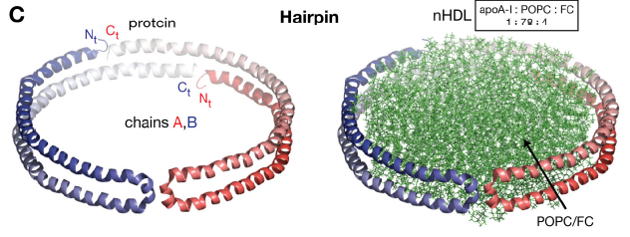 Figura 3C. Modelo horquilla para pelo (Hairpin): en este modelo las dos cadenas de ApoA-I en vez de asociarse de manera antiparalela, forman una semicircunferencia asociadas únicamente de manera intercatenaria por un pequeño grupo
de aminoácidos.
Figura 3C. Modelo horquilla para pelo (Hairpin): en este modelo las dos cadenas de ApoA-I en vez de asociarse de manera antiparalela, forman una semicircunferencia asociadas únicamente de manera intercatenaria por un pequeño grupo
de aminoácidos.
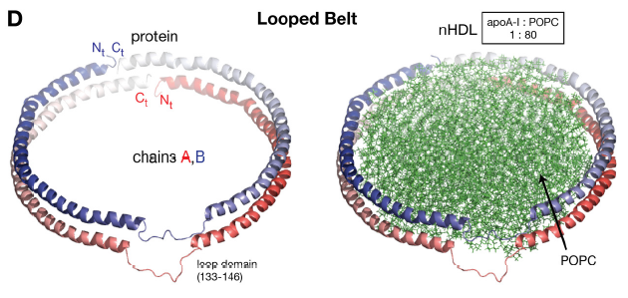 Figura 3D. Modelo cinturón con bucle (Looped Belt): en este modelo se produce una variación del modelo del cinturón doble a partir de estudios que determinaban que ciertas regiones de la ApoA-I eran altamente dinámicas, y presentaban
un plegamiento aleatorio.
Figura 3D. Modelo cinturón con bucle (Looped Belt): en este modelo se produce una variación del modelo del cinturón doble a partir de estudios que determinaban que ciertas regiones de la ApoA-I eran altamente dinámicas, y presentaban
un plegamiento aleatorio.
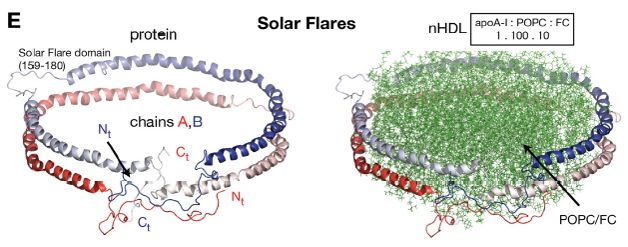 Figura 3E. Modelo ráfagas solares (Solar Flares): este modelo se desarrolló a partir del anterior incorporando información de nuevos estudios, en este las cadenas se disponen de manera antiparalela como en el modelo doble cinturón.
Fuente de la figura 3, Gogonea, 2016.
Figura 3E. Modelo ráfagas solares (Solar Flares): este modelo se desarrolló a partir del anterior incorporando información de nuevos estudios, en este las cadenas se disponen de manera antiparalela como en el modelo doble cinturón.
Fuente de la figura 3, Gogonea, 2016.
A pesar de que todos estos modelos incorporan datos experimentales de estudios biofísicos, a día de hoy la conformación de la ApoA-I se encuentra todavía en debate.
HDL MADURAS
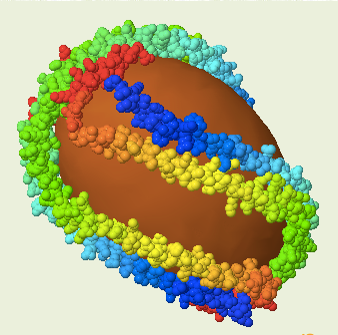
Cuando las HDL van captando lípidos de los tejidos extrahepáticos se transforman en las HDL maduras, cuyo núcleo lipídico tiene una forma más elipsoidal. En este modelo aparecen 362 fosfolípidos y 4 cadenas de ApoA-I, representado en el modelo inferior
obtenido de la página Biomodel.
Modelo estructural de las HDL maduras.
ESTRUCTURA DE LA APOA-I
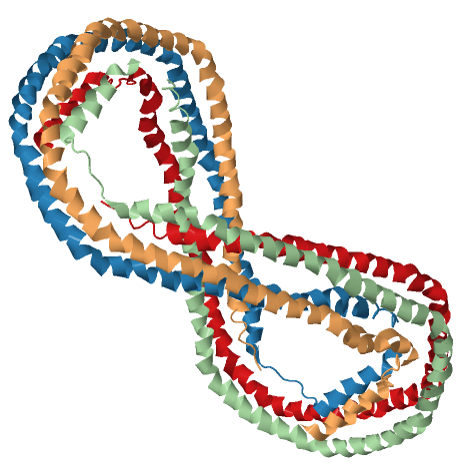
Se han realizado gran cantidad de modelos estructurales sobre la estructura de la ApoA-I en estado libre de lípidos, pero no se ha llegado a un acuerdo, ya que en este estado es muy difícil cristalizar la proteína. La estructura cuando se encuentra unida a lípidos está
formada por 4 moléculas de la apoproteína asociadas a través de sus caras hidrofóbicas, según se observa en el siguiente modelo, en el que cada cadena polipeptídica se muestra en un color diferente.
Estructura de la APOA-I asociada a lípidos.
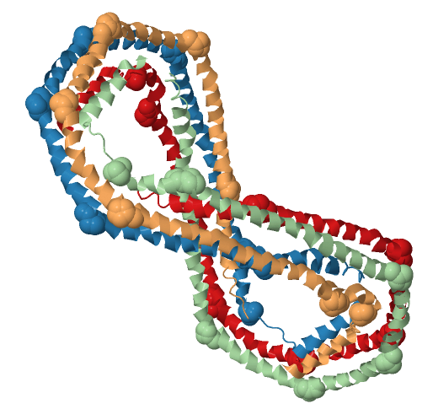
La molécula se compone casi en su totalidad de una alfa-hélice anfipática pseudo-continua (en todas las cadenas el porcentaje de alfa hélice se encuentra en torno a 85-87%), que está puntuada por torsiones en residuos
de prolina espaciados regularmente (marcados en spacefill en el modelo mostrado más abajo). Estas prolinas son las que permiten a la molécula adopte una forma de herradura.
Estructura de la APOA-I con las prolinas marcadas en estilo “spacefill”.
Las 4 cadenas se asocian entre sí formando una estructura de anillo elíptico. Este anillo se puede dividir en dos pares de moléculas A/B y C/D (ver los modelos anteriores) que están relacionadas entre sí.
Las moléculas A y B forman un dímero antiparalelo que se asocian entre sí excepto en los residuos 220-227. Esto provoca la pérdida de 4120 Å de área accesible por el solvente, de cada monómero, de manera que aparecen 8240 Å de
área enterrada (el 21% del área total de las moléculas A y B). La mitad de esta área enterrada se encuentra asociada por interacciones hidrofóbicas, y el 30% se debe a interacciones electrostáticas entre residuos cargados. Una cara
del dímero A/B es hidrofílica y la otra hidrofóbica. El exterior del tetrámero es hidrofílico mientras que el interior es hidrofóbico para permitir la captación de lípidos.
ABCA1 Y ENFERMEDAD DE TANGIER
ABCA1 es un transportador dependiente de ATP (ABC, del inglés “ATP-binding cassette”) perteneciente a la subfamilia A, que media el flujo de fosfolípidos y colesterol hacia el exterior de la célula. Esta proteína interacciona con el aceptor extracelular
ApoA-I de las lipoproteínas para generar lipoproteínas de alta densidad (HDL). ABCA1 está formada por una única cadena de 2261 aminoácidos y tiene una altura total de unos 200Å. Está constituida por distintos dominios con diferentes
funciones dentro de la proteína que se resumen a continuación y se muestran en el siguiente modelo:
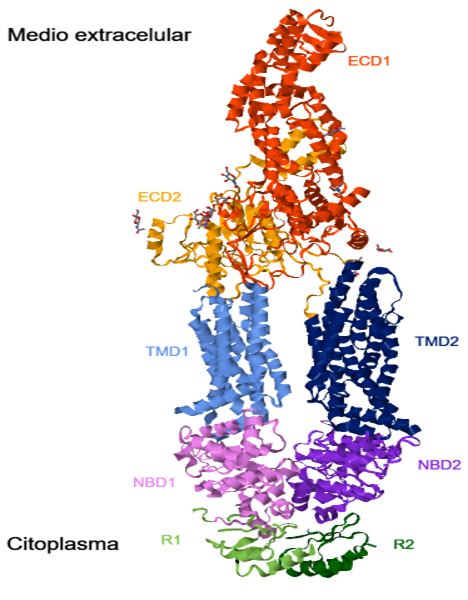
Dominios de ABCA1
● Dominios transmembrana (TMD1 y TMD2, en el modelo de color azul) Formados por seis hélices transmembrana cada uno y plegados de manera independiente.
Son los responsables de la translocación del sustrato a través de la bicapa lipídica, proceso dependiente de ATP y que necesita una comunicación alostérica entre los dominios
TMDs y
NBDs. Se ha propuesto un mecanismo de acceso lateral por la hemimembrana interna, seguido de un movimiento de “flip-flop” por los fosfolípidos dependiente de ATP.
● Dominios de unión a nucleótidos (NBD1 y NBD2, en el modelo de color rosa y morado respectivamente) Los NBDs son los dominios que permiten la unión del ATP y su transformación en ADP y Pi para que se pueda dar correctamente
la translocación de fosfolípidos y colesterol.
● Dominios reguladores (R1 y R2, en el modelo de color verde) Los dominios R1 y R2 están íntimamente unidos. Se encargan de modular la actividad transportadora, de estabilizar las interacciones entre NBD1 y NBD2 y además parece
que mejora la cooperatividad entre las dos mitades que conforman esta proteína.
● Dos dominios extracelulares (ECD1 y ECD2, en el modelo de color rojo y naranja respectivamente)
Ambos ECDs se encuentran plegados de forma retorcida uno sobre otro. Estos dominios forman una estructura que puede ser dividida en tres partes:
○ La base está constituida por dos secuencias en conformación α/β de ambos dominios (ECD1 y ECD2).
○ El túnel hidrofóbico está formado principalmente por α-hélices en ECD1 y una horquilla de α-hélices en ECD2. Sirve de paso y almacén temporal de los lípidos que van a formar las HDL. Existe un gran cambio conformacional
en el momento en el que los lípidos pasan de la membrana al ECD.
○ La tapa colocada sobre el túnel, está constituido por un dominio α/β de ECD1.
Se ha sugerido que estos grandes dominios de la proteína son los responsables de la unión a ApoA-I. No obstante, se necesita información adicional para definir la función biológica de estos dominios ECDs.
En total, se han observado 97 mutaciones de cambio de sentido en ABCA1 de humanos que están asociadas a enfermedades como la enfermedad de Tangier y deficiencia de HDL familiar. La enfermedad de Tangier es una enfermedad
genética rara caracterizada por una importante disminución de HDL y una alta incidencia en enfermedades cardiovasculares. Las mutaciones más estudiadas son:
● W590S en ECD1
Se encuentra situada en la zona inferior del túnel extracelular. Produce la disminución de la translocación de fosfolípidos, aunque se ha observado que la proteína
se encuentra correctamente localizada en la membrana plasmática y presenta una capacidad similar de unión a ApoA-I respecto a la proteína silvestre.
● C1477R en ECD2 Se localiza en la base de la región ECD2, cerca de la superficie extracelular de TMD1. En este caso, la proteína se expresa y transloca los fosfolípidos de manera
correcta; sin embargo, la unión a ApoA-I disminuye. Este hecho sugiere que ABCA1 junto con la bicapa lipídica unen de manera cooperativa apoA1 para formar HDL, aunque no se sabe aún si esto es así realmente.
En el siguiente modelo se muestra la localización de los residuos W590 y C1477 en modelo “spacefill”:
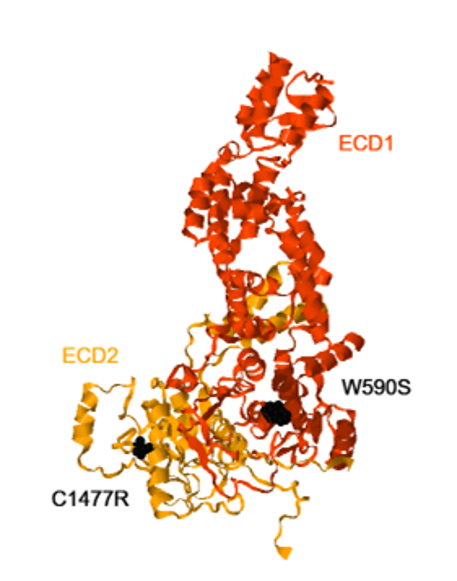
Mutaciones relacionadas con la enfermedad de Tangier
SR-BI
El receptor SR-BI, es una proteína receptora de membrana multiligando que funciona como un receptor de lipoproteína de alta densidad (HDL) cuya función principal es facilitar la entrega de colesterol de las HDL a las células, y por lo tanto es una proteína
importante responsable de la eliminación del colesterol.
La interacción de las lipoproteínas de alta densidad (HDL) con su receptor (SR-BI) es fundamental para reducir los niveles de colesterol en plasma y reducir el riesgo de enfermedad cardiovascular.
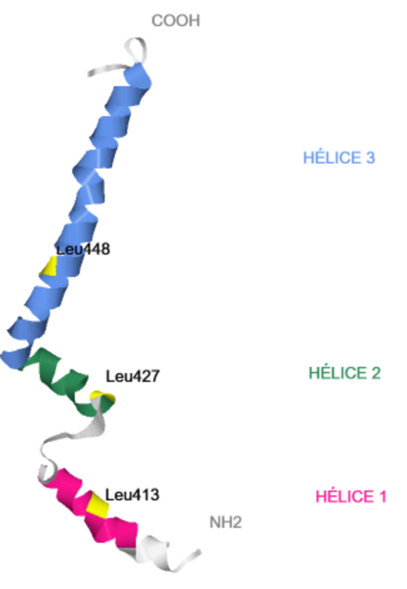
En el siguiente enlace se muestran los aspectos estructurales de la región C-terminal transmembranal (residuos 405 a 475).
Estructura del dominio transmembrana C-terminal de SR-BI
Esta región de SR-BI alberga un motivo de dimerización de cremallera de leucina, que cuando muta altera la capacidad del receptor para unirse a HDL y mediar la entrega de colesterol. Se ha comprobado que la mutación de varios
residuos de la cremallera de leucina interrumpe la función SR-BI y estas pérdidas en la función se relacionan con la incapacidad de SR-BI para formar dímeros, de lo que se ha deducido que la función del receptor probablemente está
mediada por la dimerización de éste.
Se ha examinado la importancia funcional de cada leucina individual dentro de este motivo de SR-BI, y se ha comprendido mejor por qué varias mutaciones de leucina individuales conducen a la interrupción de las diferentes funciones
del receptor. Los tres residuos mutados se muestran en amarillo en el modelo anterior.
● La mutación de L413 altera significativamente la unión de HDL.
● L413A, L427A y L448A mostraron disminuciones en varias funciones de transporte de colesterol mediadas por SR-BI.
También se han identificado regiones yuxtamembranas del dominio extracelular de SR-BI que pueden interactuar con la superficie lipídica para facilitar las funciones de transporte de colesterol del receptor. Para facilitar
este transporte, HDL y SR-BI deben formar un "complejo productivo" mediante el cual HDL se une al dominio extracelular de SR-BI en una conformación precisa que permite el transporte eficiente de lípidos.
La estructura cristalina de alta resolución ha proporcionado información valiosa sobre la estructura del dominio extracelular de SR-BI así como de su función, sin embargo, no está disponible una estructura completa de alta resolución
de SR-BI, lo cual dificulta una comprensión de la interacción HDL/SR-BI.
ASPECTOS BIOTECNOLÓGICOS: NANODISCOS
Los nanodiscos son unas estructuras moleculares artificiales de pequeño tamaño que están compuestas por un pequeño disco de lípidos lo suficientemente grande como para contener una copia de una proteína de interés rodeada por un cinturón estabilizador
de proteínas. Los nanodiscos fueron diseñados buscando inspiración en la naturaleza, en concreto, utilizando como referencia lipoproteínas del tipo de las HDL.
Los nanodiscos resultan muy importantes para los investigadores que tratan de determinar la estructura de proteínas de membrana que tradicionalmente han sido muy difíciles de estudiar porque necesitan protegerse en un entorno
lipídico, es decir, con características hidrofóbicas y apolares. Los nanodiscos se diseñan a partir de apolipoproteínas naturales, cambiando la longitud de sus cadenas hasta alcanzar el tamaño que permita rodear el disco lipídico deseado.
Las proteínas diseñadas se denominan MSP (membrane scaffolding proteins) y, al igual que las apolipoproteínas, forman una estructura alfa-helicoidal en la que los
aminoácidos hidrofóbicos (en rojo) se disponen preferentemente hacia la superficie interna del anillo, con el fin de estabilizar las porciones hidrofóbicas de los lípidos en la membrana. Por el contrario, los
aminoácidos hidrofílicos (en azul), se disponen mayormente hacia el exterior del anillo, en contacto con el medio acuoso polar.

Cinturón de proteínas del nanodisco
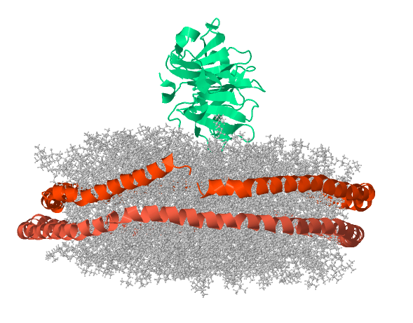
A continuación, a modo de ejemplo, se muestra la estructura del
dominio de unión a membrana (en verde) de MT1-MMP (metaloproteinasa de matriz de membrana tipo 1) embebida en un nanodisco. Se distinguen las
proteínas (en tonos anaranjados) y los lípidos (en gris).
Dominio de unión a membrana de MT1-MMP unido a un nanodisco
Guion elaborado por Irene Bragado García, Ana Conde Yáñez, Nel Marín Sánchez y Raúl Mateos Pablos, alumnos de Química e Ingeniería de Proteínas, 3er curso del Grado en Biotecnología, curso 2019/20.
Cuestiones
Responder al cuestionario correspondiente en la página de Studium
Referencias
Biomodel, http://biomodel.uah.es/
Borhani, D.W., Rogers, D.P., Engler, J.A. & Brouillette, C.G. (1997) Crystal structure of truncated human apolipoprotein A-I suggests a lipid-bound conformation, Proc Natl Acad Sci USA 94, 12291–12296. doi: 10.1073/pnas.94.23.12291
Denisov, I.G. & Sligar, S.G. (2016) Nanodiscs for structural and functional studies of membrane proteins. Nature Structural and Molecular Biology 6, 481-486. doi:
Chadwick, A.C., Jensen, D.R., Hanson, P.J., Lange, P.T., Proudfoot, S.C., Peterson, F.C., Volkman, B.F. & Sahoo, D. (2017). NMR Structure of the C-Terminal Transmembrane Domain of the HDL Receptor, SR-BI, and a Functionally
Relevant Leucine Zipper Motif. DOI: 10.1016/j.str.2017.01.001
Gogonea, V. (2016) Structural Insights into High Density Lipoprotein: Old Models and New Facts. Front. Pharmacol. 6:318.
doi: 10.3389/fphar.2015.00318
Goodsell D., RCSB PDB Molecule of the Month doi: 10.2210/rcsb_pdb/mom_2019_9
Marcink, T.C., Simoncic, J.A., An, B., Knapinska, A.M., Fulcher, Y.G., Akkaladevi, N., Fields, G.B. & Van Doren, S.R. (2019) MT1-MMP Binds Membranes by Opposite Tips of Its beta Propeller to Position It for Pericellular Proteolysis.
Structure 27: 281-292.e6. doi:10.1016/j.str.2018.10.008
Lehninger, A.L.; Nelson, D.L. & Cox, M.M. (2009) Principios de Bioquímica, 5ª ed. Omega, Barcelona
PDB www.rcsb.org, H.M. Berman, J. Westbrook, Z. Feng, G. Gilliland, T.N. Bhat, H. Weissig, I.N. Shindyalov, P.E. Bourne (2000) The Protein Data Bank URL: Nucleic Acids Research, 28: 235-242.
Phillips MC. (2018) Is ABCA1 a lipid transfer protein?, JLR Papers in Press 749-763. doi:
10.1194/jlr.R082313
Qian H., Zhao X., Cao P., Lei J., Yan N. & Gong X. (2017) Structure of the Human Lipid Exporter ABCA1, Cell 169: 1228-1239.e10. doi:
10.1016/j.cell.2017.05.020