Introducción
La aconitasa y la IRP-1 (Iron Regulatory Protein-1) son dos proteínas aparentemente muy diferentes. La primera es una enzima que cataliza la reacción de isomerización del citrato en isocitrato. La segunda regula el metabolismo del
hierro uniéndose a distintos mRNA según las concentraciones de este metal, lo que resultará en cambios en su almacenamiento y liberación.
Sin embargo, el estudio de las secuencias de aminoácidos de ambas proteínas ha llevado a la conclusión de que las dos presentan la misma cadena polipeptídica. Estas dos moléculas, aunque llevan a cabo acciones diferentes, no son
más que dos formas prácticamente idénticas de la misma proteína, que con apenas unos cambios son capaces de participar en dos procesos metabólicos que nada tienen que ver entre sí.
Las proteínas que presentan esta capacidad de llevar a cabo dos funciones distintas se denominan comúnmente en inglés “moonlight proteins”.
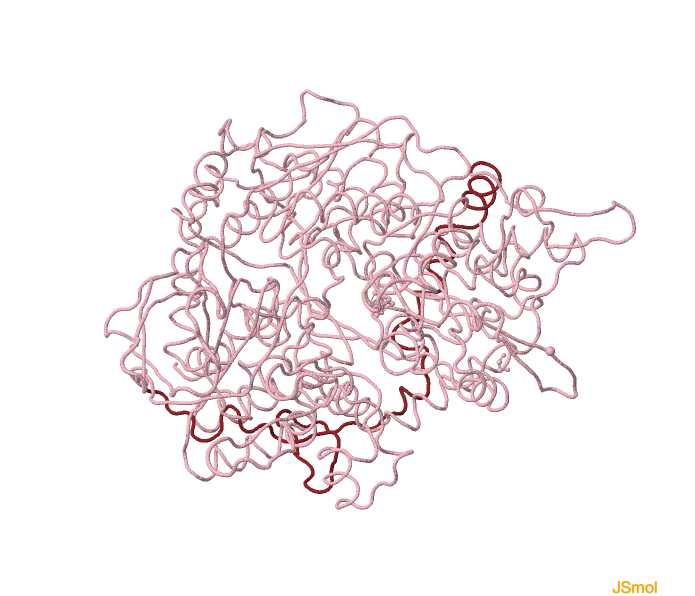
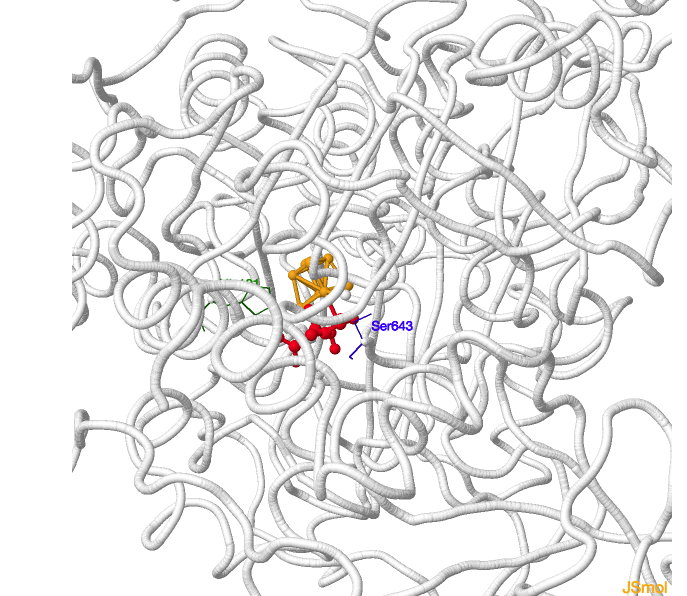
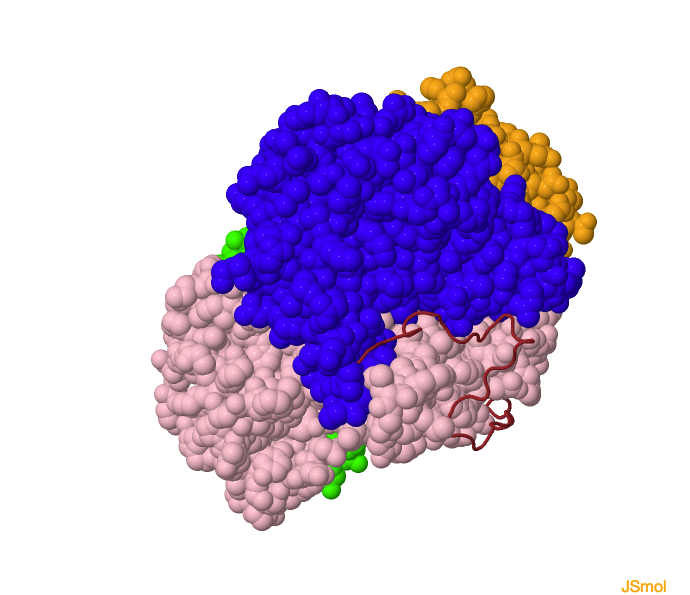
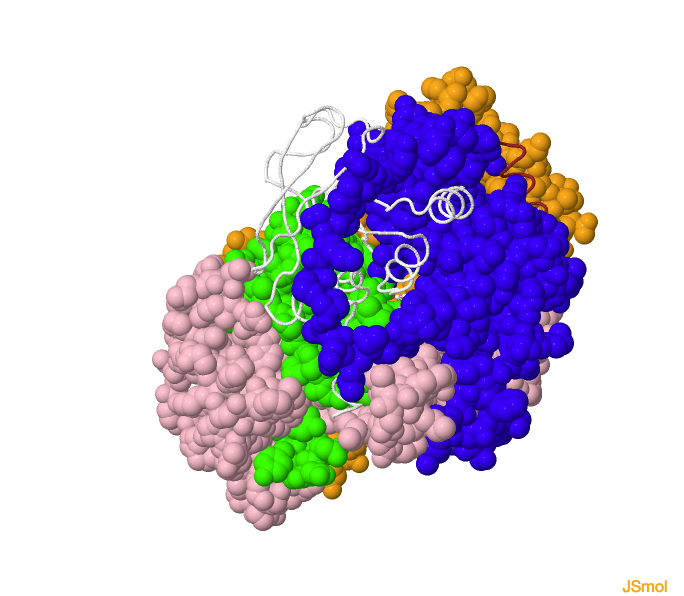
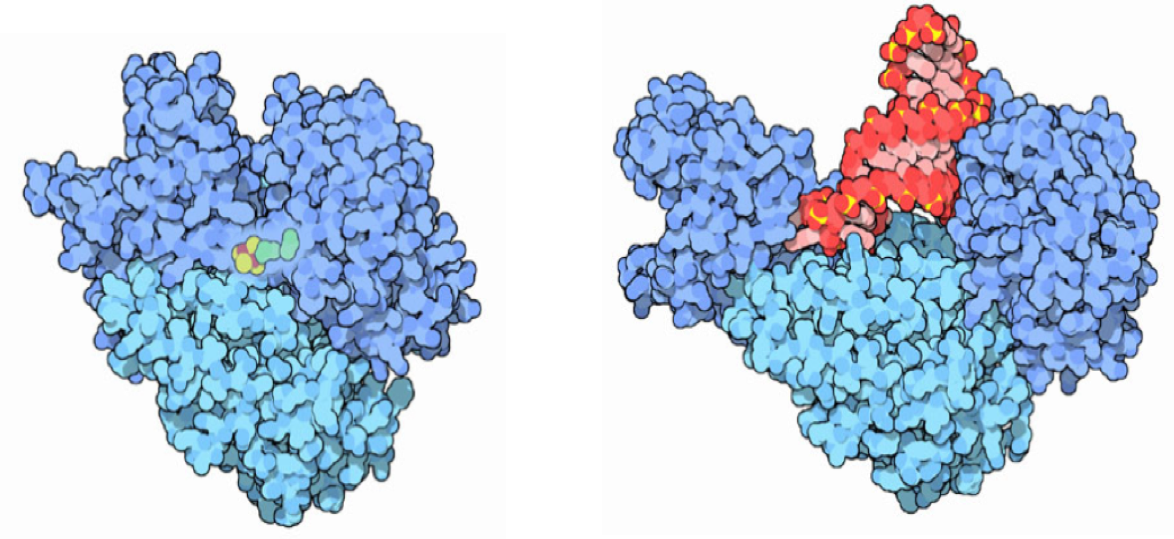 Figura 1. Representación de la estructura de la aconitasa (izquierda) y de IRP1(derecha)
Figura 1. Representación de la estructura de la aconitasa (izquierda) y de IRP1(derecha)
DUALIDAD FUNCIONAL:
Es por tanto evidente que para que la misma proteína ejerza dos funciones distintas, ésta ha de sufrir ciertos cambios conformacionales que le permitan la transición de una función a otra.
La estructura de la proteína se compone de cuatro dominios que se presentan en el siguiente orden: dominios 4, 1, 2 y 3. Además, entre los dominios 3 y 4 existe una estructura proteica que varía de una a otra conformación funcional,
y que se denomina linker joining.
La estructura de la proteína como IRP1 muestra una conformación 25 Å más abierta que la de la aconitasa citosólica (c-aconitasa). De hecho, la estructura cristalizada de la c-aconitasa muestra una proteína más compacta, donde
los residuos implicados en la función de la proteína como IRP1 están “enterrados” e inaccesibles. Por tanto, los accesos de la proteína son diferentes para cada conformación en función de la unión de sus respectivos ligandos.
La proteína ejerce su función como aconitasa cuando se le une un centro ferro-sulfurado (4Fe-4S), y es precisamente la unión o no de este centro lo que parece actuar como mecanismo para regular la actividad de IRP1, es decir,
es lo que permite el intercambio entre las dos conformaciones funcionales. Esto es así porque fenómenos como la unión del núcleo ferro-sulfurado impiden la unión del enzima al RNA, evitando así su función como IRP1.
En cuanto a los cambios conformacionales, los más importantes son los que afectan a los dominios 3 y 4 de la proteína.
Por un lado, el dominio 4 sufre una rotación de 32º y una traslación de unos 14 Å. Este cambio elimina las interacciones entre los dominios 3 y 4 que se producen en la aconitasa y favorece la formación de nuevas interacciones
en los dos sitios de unión a RNA presentes en IRP1. Asimismo, dado que el dominio 4 interacciona con el dominio 1 por medio de uniones hidrofóbicas, los residuos 90 a 170 del dominio 1 se mueven junto con el dominio 4 durante estos
cambios conformacionales. Por otro lado, el dominio 3 sufre una rotación de 52º y una traslación de aproximadamente 13 Å.
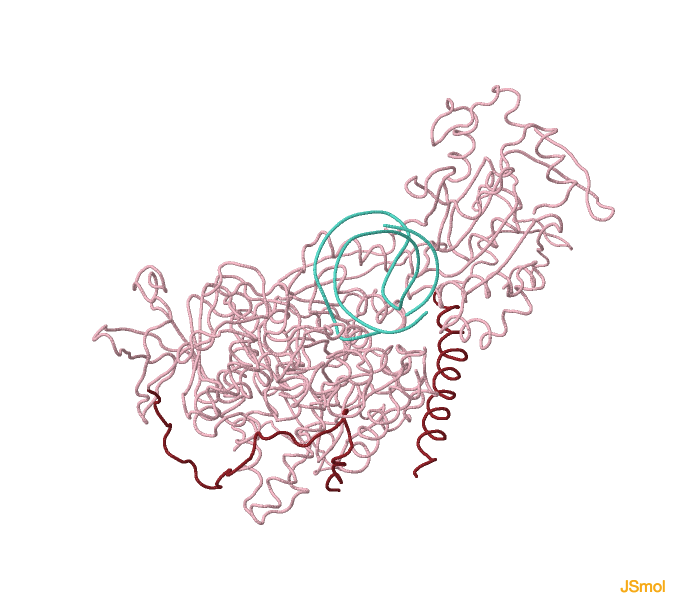
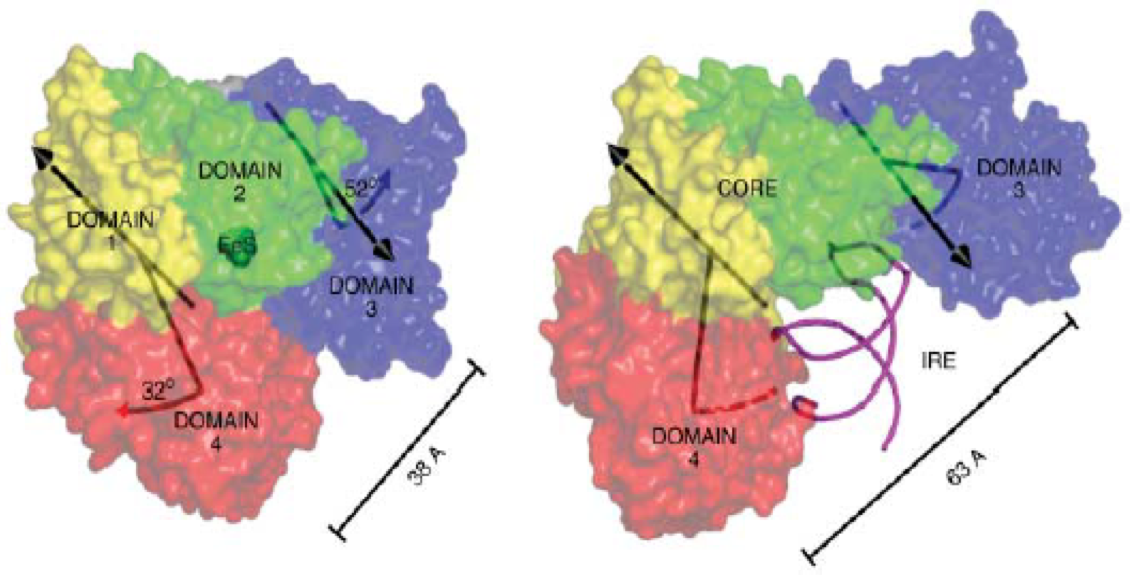 Figura 2. Diferencias en la posición de los dominios entre c-aconitasa (izquierda) y complejo IRP1: IRE-RNA (derecha)
Figura 2. Diferencias en la posición de los dominios entre c-aconitasa (izquierda) y complejo IRP1: IRE-RNA (derecha)
Un cambio conformacional en el linker joining entre los dominios 3 y 4 es fundamental para su reposicionamiento respecto del núcleo proteico formado por los dominios 1 y 2. En la c-aconitasa, los residuos 593 a 614 en esta zona comprenden
dos hélices α (21 y 22)
(en color marrón) separadas por un residuo de prolina. En el caso de IRP1, se forma
una única hélice α que implica también los residuos 593 a 614.
Estructura del linker joining en la aconitasa citosólica.
Estructura del linker joining en IRP1.
ACONITASA CITOSÓLICA
Aconitasa: reacción catalizada
La aconitasa es una proteína que pertenece a la familia de las hidrolasas dependientes de un centro ferro-sulfurado que se encargan de la catálisis de la reacción de isomerización de β-hidroxiácidos.
En concreto, la aconitasa cataliza la isomerización del citrato en isocitrato a través de una deshidratación y una hidratación sucesivas, formándose un intermediario, el cis-aconitato, entre ambos procesos.
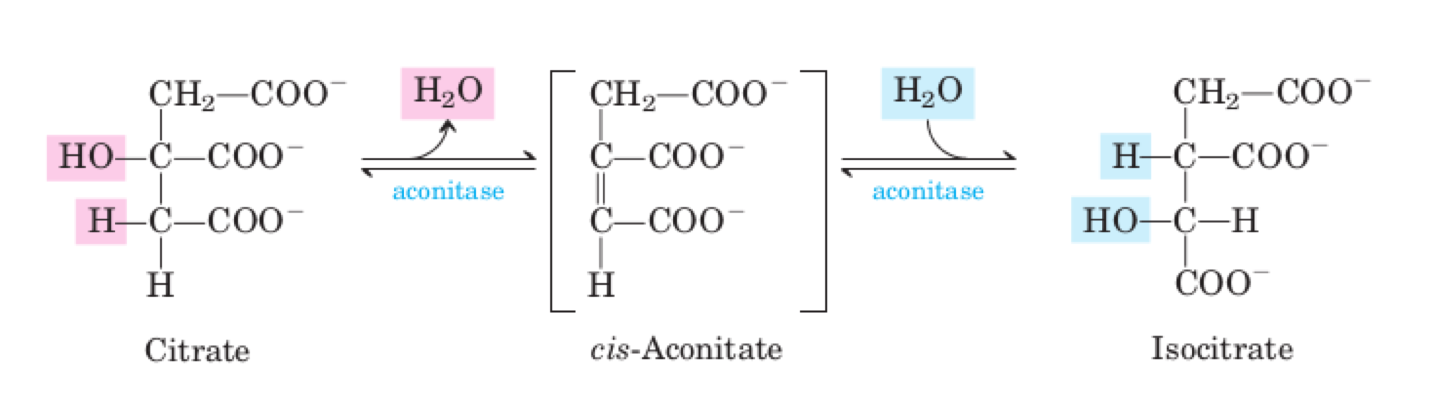 Figura 3. Esquema de la reacción catalizada por la aconitasa
Figura 3. Esquema de la reacción catalizada por la aconitasa
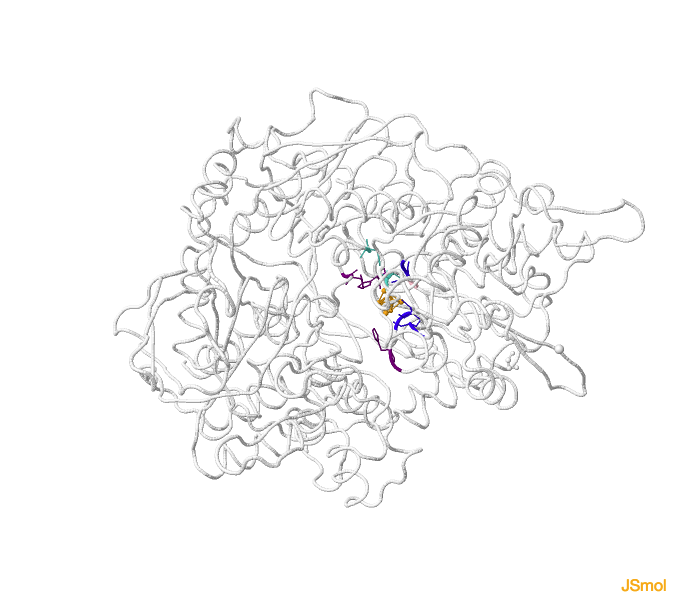
El sustrato se estabiliza en el centro activo de la enzima gracias a interacciones electrostáticas con el centro ferro-sulfurado, que además lo ayuda a colocarse en la posición correcta para interactuar con los residuos catalíticos. Este
cluster de Fe-S (en color naranja) se une a la proteína por medio de los residuos
Cys437, Cys503 y Cys506 (en color azul oscuro).
Por otro lado, residuos de los cuatro dominios que conforman la proteína forman parte del centro activo. Algunos de estos aminoácidos son
histidinas (en color morado),
asparaginas (en color azul claro) y
serinas (en color rosa). La presencia de estos aminoácidos en el centro activo es importante, pues permite crear un microentorno especializado en el que suceda la reacción.
Centro activo de la aconitasa.
Por tanto, en el centro activo del enzima existen aminoácidos responsables de la unión y el anclaje del sustrato al enzima, a lo que también contribuye el centro ferro-sulfurado, y, por otro lado, tenemos aminoácidos encargados de llevar a cabo la catálisis
propiamente dicha.
La
Ser642 es capaz de aceptar el protón que cede el sustrato actuando como base, gracias al entorno químico en que se encuentra. Por otro lado, la
His101 actúa como un ácido donando su protón para la reacción.
En primer lugar, ocurre una deshidratación: la His101 proporciona un H+ que formará H2O con un OH- del C3 del sustrato. Después, la Ser642 actuará como base y captará un H+ del C2 del
sustrato, incorporándolo a su cadena lateral. Así se forma el intermediario cis-aconitato, con un doble enlace entre el C2 y el C3.
Los residuos Ser642 e His101 son importantes para el mecanismo catalítico.
A continuación, el cis-aconitato sufre un giro de 180º y comienza la reacción correspondiente de hidratación. La His101 vuelve a captar un átomo de H+, de la molécula de H2O generada anteriormente, y el OH- restante se vuelve a unir al sustrato, aunque esta vez en una posición distinta. La Ser642 cede el H+ de vuelta al sustrato y este se libera en forma ya de isocitrato, completándose la reacción.
Aconitasa mitocondrial
La aconitasa citosólica y la aconitasa mitocondrial son dos isoenzimas que pertenecen a la misma familia. Como isoenzimas, ambas catalizan la misma reacción química, pero difieren en su secuencia aminoacídica y, como consecuencia,
en su estructura tridimensional. Cabe destacar que la única que puede actuar como IRP-1 es la aconitasa citosólica, y no la mitocondrial.
La aconitasa citosólica comprende una secuencia de 889 aminoácidos de longitud, mientras que la mitocondrial solamente presenta 754. Los dominios, por tanto, presentan configuraciones diferentes:
Dominios de la aconitasa citosólica.
Dominios de la aconitasa mitocondrial.
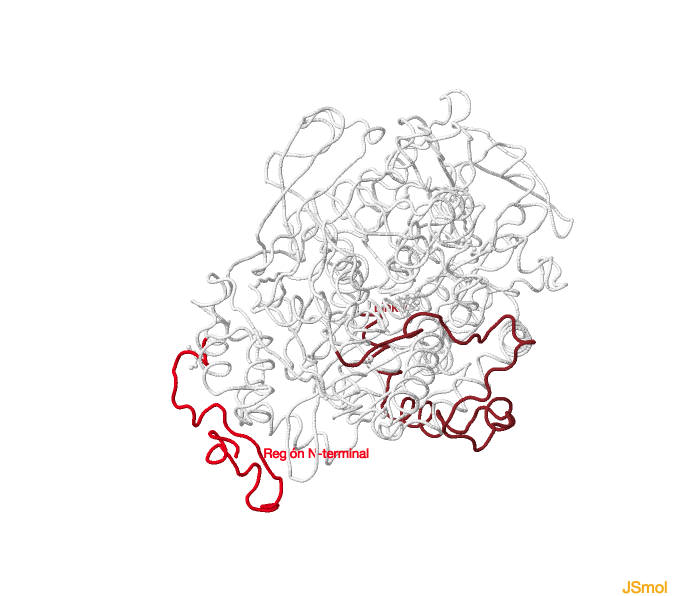
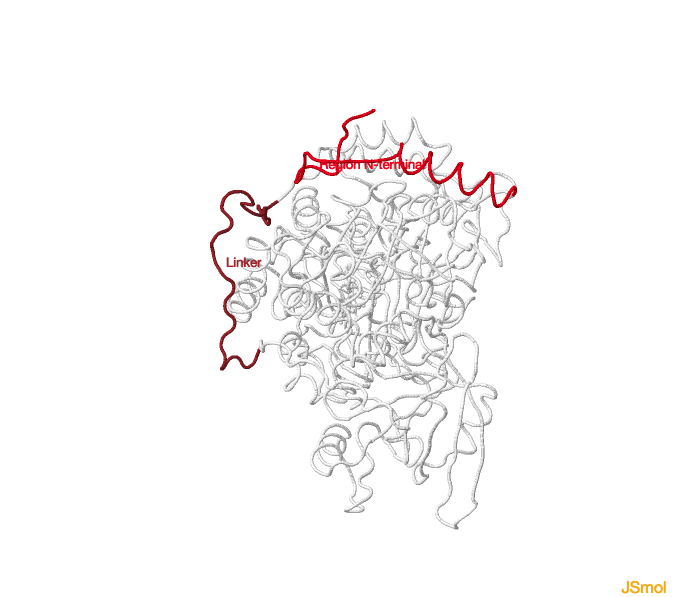
En el dominio 1, se observa un plegamiento diferente para la región N-terminal (residuos del 1 al 33) al comparar las dos proteínas. El linker joining en la aconitasa citosólica (593 a 654) es más largo que en la mitocondrial (513 a 536), y además se
encuentra más estructurado.
Región N-terminal y linker joining de la aconitasa citosólica.
Región N-terminal y linker joining de la aconitasa mitocondrial.
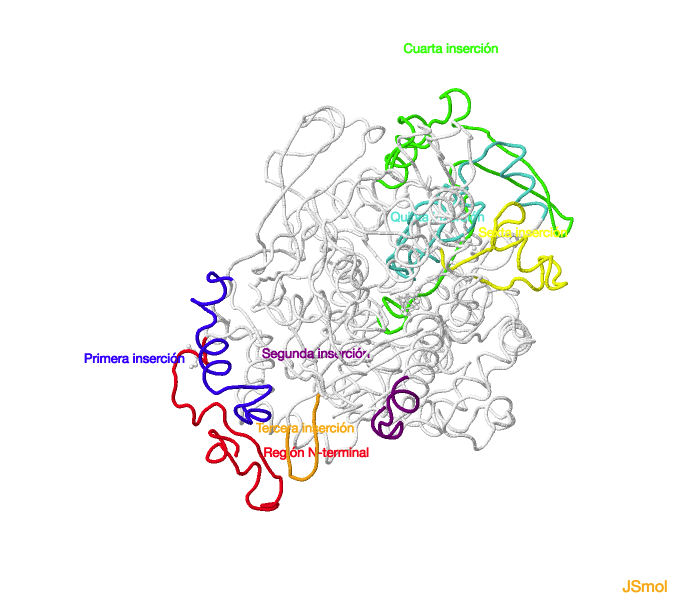
Además, cuando se compara con el isozima mitocondrial, en la aconitasa citosólica aparecen inserciones en diferentes dominios: tres en el dominio 1, dos en el dominio 3 y una en el dominio 4 otra más, siendo esta última la más grande. Al darse todas estas
inserciones, las estructuras tridimensionales de estas dos isoenzimas son significativamente distintas.
Inserciones en la estructura de la aconitasa citosólica.
IRP-1s
Iron Regulatory Proteins
El hierro es un elemento esencial para los seres humanos, pero puede llegar a ser tóxico en concentraciones elevadas, por eso es necesario un complejo sistema de regulación. La homeostasis del hierro en la célula animal está controlada
post-transcripcionalmente por las proteínas reguladoras de hierro o Iron Regulatory Proteins, IRP1 e IRP2. En situaciones de escasez de hierro o estrés oxidativo, las IRPs modulan la expresión de genes relacionados con el metabolismo
del hierro. Para ello, se unen a estructuras en bucle del mRNA formadas por unos 30 nucleótidos, denominadas elementos de respuesta a hierro (IRE) para modular la expresión de dichos genes. Ya se han identificado más de 150
IRE, que controlan la expresión de proteínas como la ferritina, la ferredoxina, el receptor de transferrina, etc.
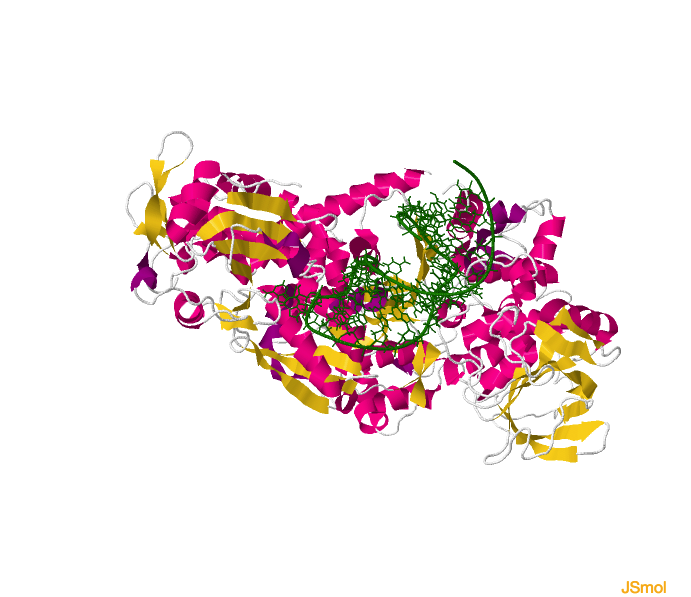
IRP1 unido a IRE del mRNA del receptor de transferrina.
Esta regulación depende de la posición del IRE en el mRNA. Si la IRP se une en el extremo 5’UTR, la traducción se reprimirá, mientras que, si el IRP se une en el extremo 3’, se favorecerá la traducción indirectamente, ya que se bloquea la degradación
del mRNA.
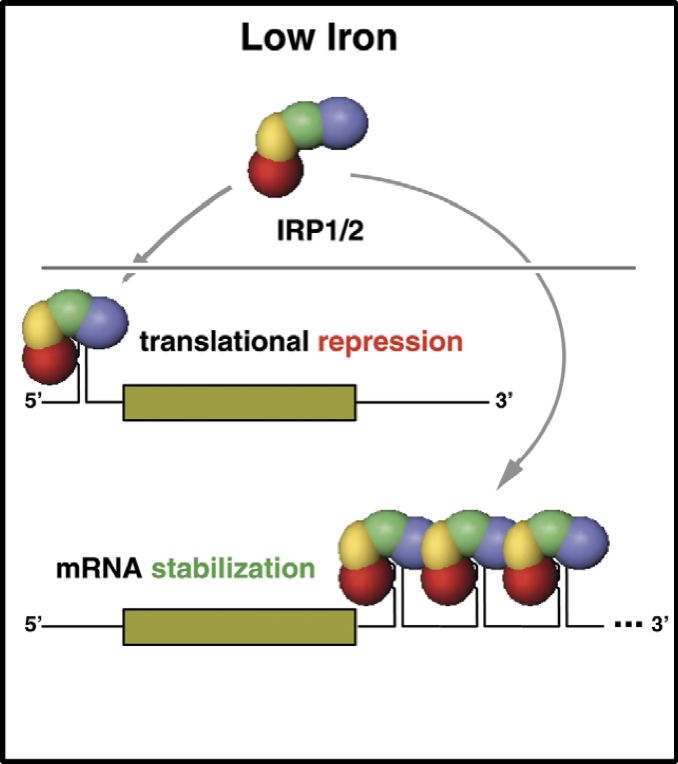 Figura 4. Regulación de la expresión según la posición del IRE
Figura 4. Regulación de la expresión según la posición del IRE
Iron Responsive Elements (IRE)
La estructura típica de un IRE, formado por 30 nucleótidos, es un
bucle apical (en rojo) de seis nucleótidos. Este bucle se encuentra sobre una doble
hélice superior (en azul) semirrígida de cinco pares de bases, que a su vez se apoya en una
hélice intermedia (en verde) de unión y, este, sobre una
hélice inferior (en morado) de longitud variable.
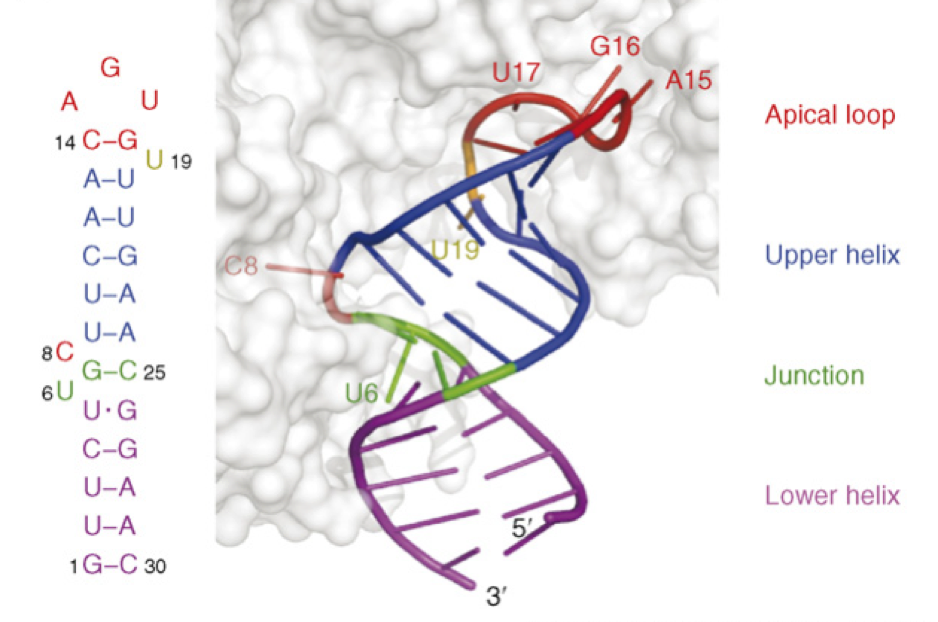 figura 5: Estructura del IRE del mRNA de la ferritina H
figura 5: Estructura del IRE del mRNA de la ferritina H
En todos los IRE podemos encontrar una secuencia de seis bases CAGUGX, siendo X uracilo, citosina o adenina, que forman el bucle apical. En la parte apical del bucle encontramos un triplete AGU, conservado en todos los IRE. Otro elemento conservado es
una citosina no apareada en la posición 8 entre la hélice intermedia y la superior.
Unión IRP-1: IRE
Como ya hemos visto, la estructura de la IRP-1 es de unos 25 Å más ancha que la de la aconitasa citosólica, formando una “L”, ya que los dominios 3 y 4 se extienden perpendicularmente al núcleo de la proteína. Esta conformación le permite interaccionar
con el IRE en dos puntos: uno con la C8, y otro con el bucle apical. En concreto, el dominio 4 de la proteína gira para exponer un bolsillo donde se unirá la C8 invariable, mientras que el dominio 3 rota hasta crear una cavidad entre
él y el dominio 2 donde se acomodan los nucleótidos A15, G16 y U17 del bucle apical. Por su parte, las hélice inferior y superior aportan la selectividad y especificidad para la unión, y la hélice intermedia, aporta la flexibilidad
para que el resto de elementos se ajusten a las interacciones.
Estructura del IRE del mRNA del receptor de transferrina.
Estos sitios de unión generalmente coinciden con los sitios de unión al cluster de hierro y azufre. La utilización de los mismos aminoácidos explica la exclusividad de la función de la enzima, es decir, no puede actuar a la vez como aconitasa y como IRP.
Interacciones entre IRP1 y IRE
Ahora, vamos a entrar en detalles en las uniones que se establecen entre la proteína IRP -1 y los nucleótidos del IRE.
Como hemos comentado en la estructura el IRE, el bucle apical está formado por un triplete AGU, el cual se coloca en la cavidad formada entre los dominios 2 y 3 de la IRP1. La
A15 (en azul) y la
G16 (en verde) establecen puentes de hidrógeno específicos con
Ser371 (en azul) y la
Lys379 (en verde) respectivamente, además de contactos de Van der Waals con otros residuos expuestos en las paredes de la cavidad. Mientras que el
U17 (en morado) forma un puente con la
Arg269.
Sin embargo, la unión clave es la que establece el C8 (en negrita) del RNA, el cual, se encuentra a modo de “sándwich” entre los residuos
Arg 713 (en rojo) y
Arg 780 (en rojo), estableciendo interacciones con su grupo 3´ fosfato. Además, la base nitrogenada establece puentes de hidrógeno con Ser681, Pro682, Asp781 y Trp782 (todas en negrita).
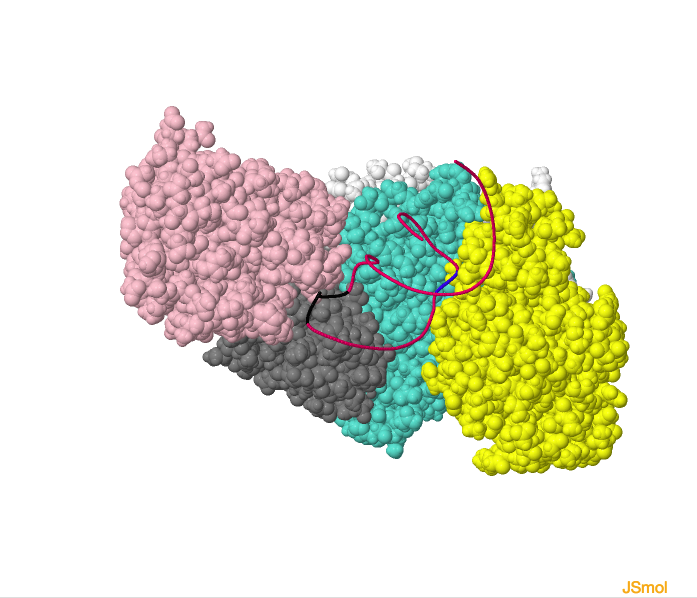
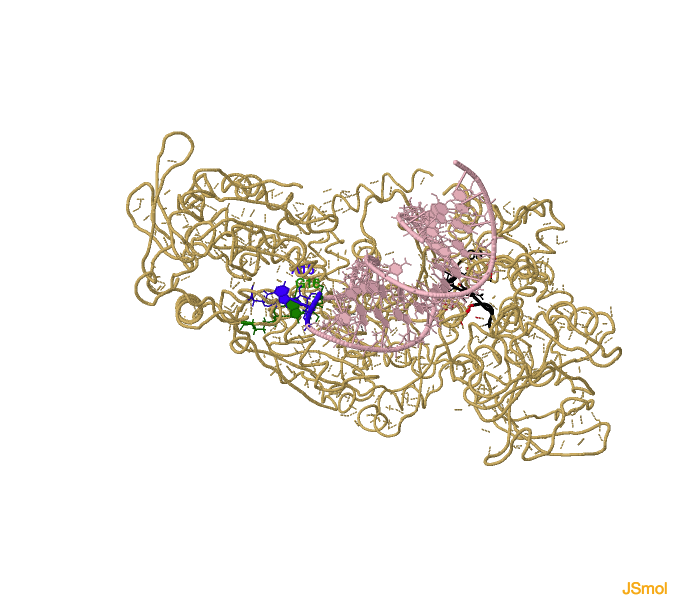
Estructura de IRP1 unido al mRNA del receptor de transferrina. Dominio 1 (en azul claro), dominio 2 (en gris), dominio 3 (en rosa), dominio 4 (en amarillo), linker (en blanco); RNA (en rojo), bucle apical (en negro) y C8 invariante (en
azul oscuro).
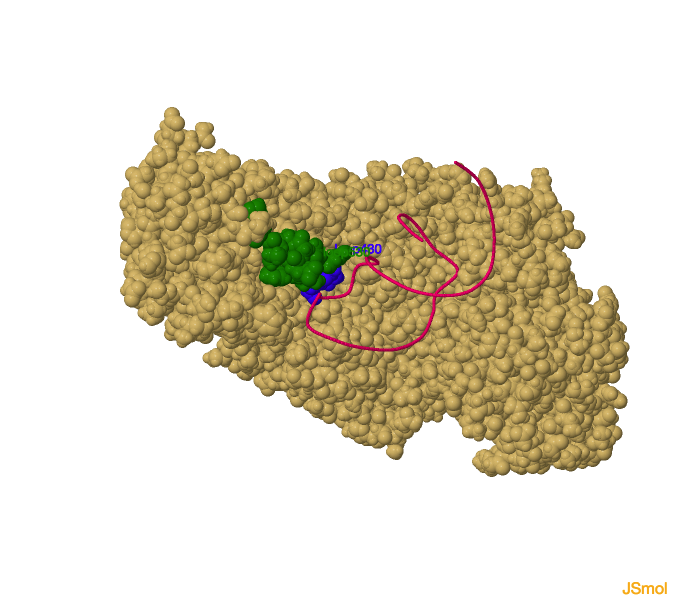
Estabilización del complejo.
Las regiones implicadas en la estabilización del complejo son los intervalos de residuos de la proteína IRP1 436-442 (denominado como región
loop 430 (en azul)) y 534-544 (denominada como
loop 530 (en verde)).
Interacciones entre IRP1 e IRE.
En lo referido al
loop 430, en la c-aconitasa el residuo Cys437, es uno de los implicado en la unión al cluster ferrosulfurado, forma parte de la hélice α 14, mientras que en la IRP1 esa hélice se extiende formando un bucle donde el residuo
437 no interviene en la unión directa al RNA, pero sí lo hacen la Thr438 y la Asn439.
En cuanto al
loop 530, un residuo importante en la estabilización de la unión al RNA es la Arg541, mediante el contacto con el 2’OH del U20.
Por último, la triple interacción entre el
loop 430, el
loop 530 y el
RNA también estabiliza el complejo, mediante la formación de cuatro puentes de hidrógeno de la Asn535: dos con la Thr438 y dos con el IRE.
Estabilización del complejo IRP1-IRE.
OTRAS MOONLIGHT PROTEINS
La aconitasa e IRP-1 son el ejemplo más utilizado para explicar las proteínas con dualidad funcional o “moonlight proteins”, pero no son las únicas que realizan dos funciones completamente distintas. Por ejemplo, la enzima retinol
deshidrogenasa (PDB 1O9J), que por un lado convierte el retinol, molécula sensible a la luz, en ácido retinoico; pero por otro lado es capaz de modificar la consistencia y la absorbancia del cristalino (lente del ojo), denominándose
en este caso eta-cristalina.
Otro ejemplo es el citocromo c (PDB 3CYT), que actúa como transportador de electrones en la mitocondria, pero cuando la célula está dañada, este sale al citosol y forma parte del apoptosoma (complejo que inicia la apoptosis).
Por último, otra moonlight protein es la Fosfoglucosa isomerasa (PDB 2PGI), que es otra enzima implicada en la glucólisis, pero también puede ser secretada al exterior de la célula y actuar como mensajero. En este último caso
puede recibir distintos nombres; como neuroleucina, factor de movilidad autocrina, mediador de la diferenciación y maduración…
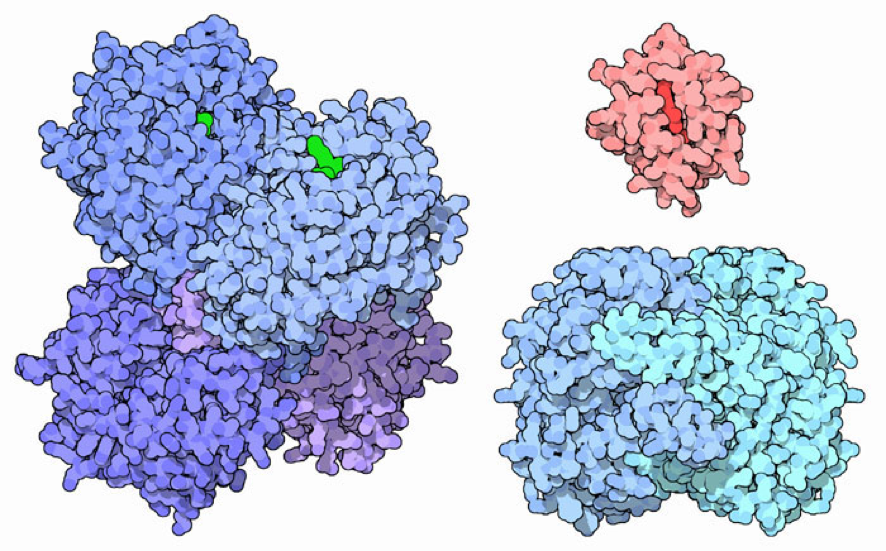 Figura 6. Otras Moonlight proteins. Enzima retinol deshidrogenasa/eta-cristalina (izquierda), citocromo c (derecha superior), y fosfoglucosa isomerasa/neuroleucina (derecha inferior).
Figura 6. Otras Moonlight proteins. Enzima retinol deshidrogenasa/eta-cristalina (izquierda), citocromo c (derecha superior), y fosfoglucosa isomerasa/neuroleucina (derecha inferior).
Guion elaborado por Mario Alcalde Redondo, Raquel Alonso Carro, Violeta Cascón Martín y Roberto García Vicente, alumnos de Química e Ingeniería de Proteínas, 3er curso del Grado en Biotecnología, curso 2017/18.
Referencias
Walden, William E., Selezneva, Anna I. et al. “Structure of Dual Function Iron Regulatory Protein 1 Complexed with Ferritin IRE-RNA”. Science, 2006, vol. 314, no 5807, p. 1903-1908.
Volz, Karl. “The functional duality ofi ron regulatory protein 1”. Current opinion in structural biology, 2008, vol. 18, no 1, p. 106-111.
Dupuy, Jérôme, Volbeda, Anne et al. “Crystal structure of human iron regulatory protein 1 as cytosolic aconitase”. Structure, 2006, vol. 14, no 1, p. 129-139.
Gruer, Megan J.; Artymiuk, Peter J. et al. “The aconitase family: three structural variations on a common theme”. Trends in biochemical sciences, 1997, vol. 22, no 1, p. 3-6.
Goodsell, D.S. “Aconitase And Iron Regulatory Protein 1”. RCSB Protein Data Bank, 2007. http://pdb101.rcsb.org/motm/89.