Introducción
En el proceso de infección vírica, la membrana celular constituye una importante barrera protectora. Los virus envueltos, como el virus de la gripe y el virus del SIDA, VIH, se encuentran rodeados de una membrana de
la misma naturaleza que la membrana plasmática que les permite fusionarse con la célula y, por ende, infectar. Este proceso se encuentra facilitado por las proteínas de fusión víricas.
Muchas de estas proteínas provienen de un precursor que se proteoliza en dos proteínas, siendo la proteína de fusión el fragmento C-terminal del precursor. Por ejemplo, la subunidad HA2 de la hemaglutinina del virus de la gripe y la proteína gp41 del virus del SIDA.
Pese a la gran heterogeneidad estructural, las proteínas de fusión se caracterizan por tener: un dominio transmembrana con extremo C-terminal, hacia el interior de la membrana vírica, para el anclaje a ésta; un dominio externo, y un péptido hidrofóbico o péptido de fusión en su extremo N-terminal, que es el que interacciona con la membrana diana.
Todas las proteínas de fusión vírica comparten un mecanismo general de acción: sufren una transición desde la conformación pre-fusión, presente en la superficie del virus antes de la interacción con la célula hasta la conformación post-fusión,
estructura en horquilla adquirida una vez se haya completado la fusión entre la membrana vírica y la membrana celular. Entre ambas conformaciones, se forma un intermediario en el que la proteína adquiere una estructura de pre-horquilla extendida que permite la fusión de membranas (Figura 1).
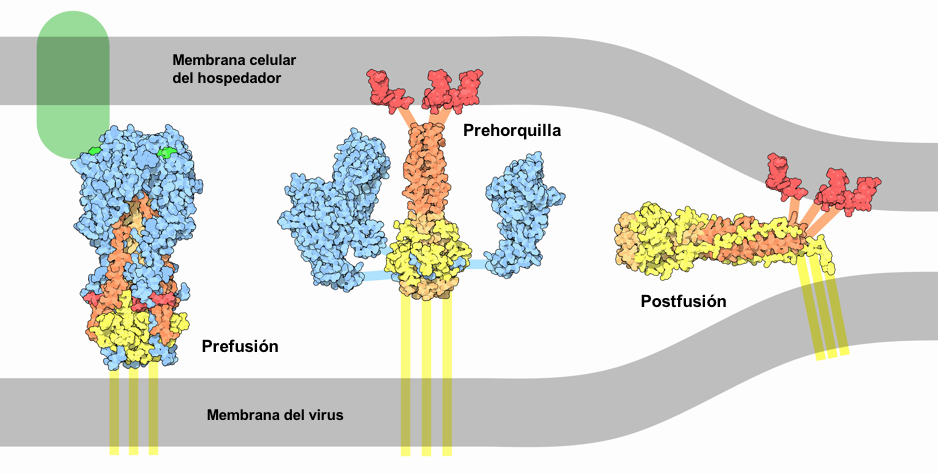 Figura 1: Esquema de las tres conformaciones de las proteínas de fusión víricas.
Figura 1: Esquema de las tres conformaciones de las proteínas de fusión víricas.
La hemaglutinina del virus de la gripe.
Los virus de la gripe son virus envueltos (es decir, con una membrana lipoproteica que rodea la nucleocápside) de RNA segmentado de cadena simple que evolucionan muy rápidamente debido a los errores de replicación y recombinación
entre segmentos génicos. Las cepas más infectivas pertenecen al virus de la gripe A y se caracterizan por tener un gran número de hospedadores. Se diferencian por el subtipo de dos de sus proteínas de la envoltura: la Hemaglutinina (HA) y la Neuraminidasa (NA). Estas dos proteínas son responsables de las interacciones de la superficie del virión con la célula hospedadora.
La Hemaglutinina (HA) desempeña dos funciones esenciales en la infección del virus de la gripe: es responsable de la unión al receptor en la superficie de la célula diana, e induce la fusión de membranas que
determina la penetración del genoma viral en la célula infectada.
HA es una glicoproteína de superficie que reconoce estructuras de polisacáridos localizados en la membrana de la célula diana presentes en glicolípidos o glicoproteínas y cuyo extremo es el ácido N-acetilneuramínico (ácido
siálico, Sia). Se considera que el enlace del ácido siálico a la penúltima galactosa del ologosacárido es esencial para la especificidad: los virus aviares se unen a 2,3-Sia y los de mamíferos a 2,6-Sia (Figura 2).
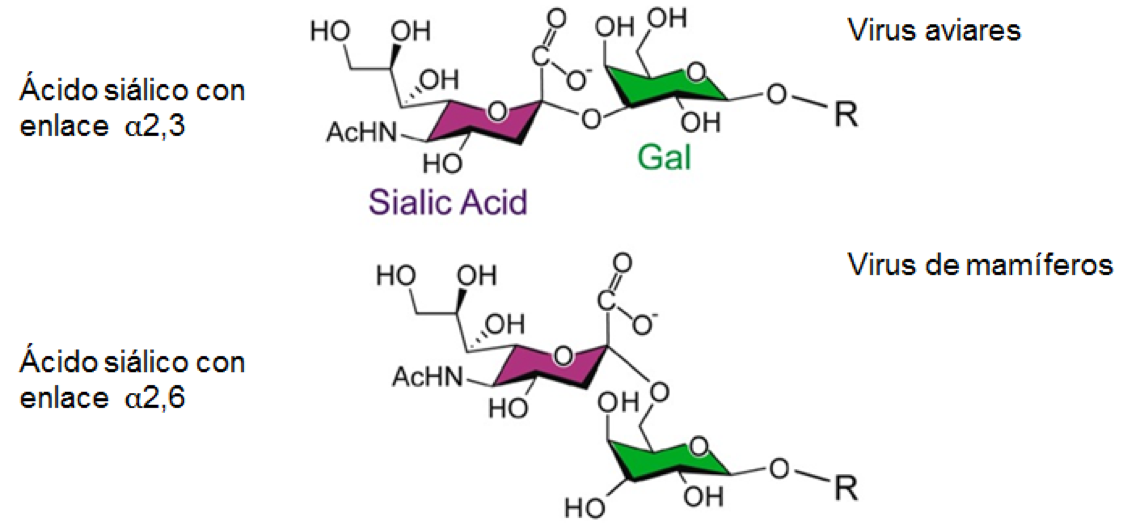 Figura 2. Reconocimiento de distintos enlaces de unión ácido siálico-galactosa según el tipo de virus.
Figura 2. Reconocimiento de distintos enlaces de unión ácido siálico-galactosa según el tipo de virus.
Síntesis de la Hemaglutinina
La proteína HA se expresa como un precursor en el retículo rugoso RER, con un péptido señal en su extremo N-terminal que se procesa durante la traducción. La proteína resultante (HA0) queda anclada a la membrana del RER
y oligomeriza formando trímeros durante la maduración. Finalmente, su procesamiento proteolítico a cargo de proteasas celulares origina dos polipéptidos independientes: HA1 y HA2, que permanecen unidos entre sí por un puente disulfuro (que va a ser esencial para el cambio conformacional que sufre la proteína en el desempeño de su función). La proteína HA2 contiene el dominio transmembrana, las secuencias heptadas repetidas HR1 y HR2 y el péptido de fusión rico en
residuos hidrofóbicos (Figura 3).
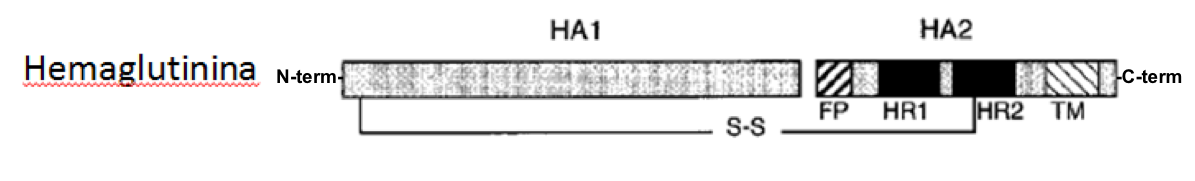 Figura 3. Esquema de la estructura general de la proteína HA (FP, péptido de fusión; HR, heptada repetida; TM, dominio transmembrana).
Figura 3. Esquema de la estructura general de la proteína HA (FP, péptido de fusión; HR, heptada repetida; TM, dominio transmembrana).
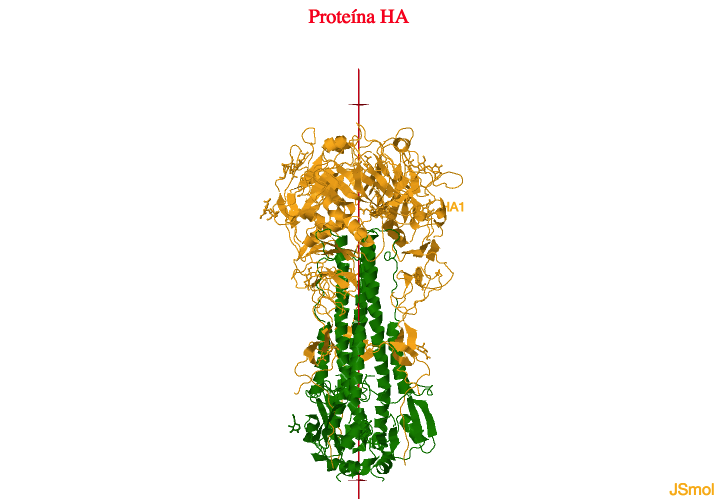
Así, la HA madura es una proteína homotrimérica que, en su forma activa, consta de dos regiones: un tallo o
HA2 (en
verde en el modelo inferior) formado por la asociación de tres hélices alfa que se enrollan entre sí adoptando una estructura de coiled-coil o “rollo enrollado”, y una región globular externa
HA1 (en
naranja), lejana a la membrana.
Estructura de la hemaglutinina del virus de la gripe.
Unión al receptor
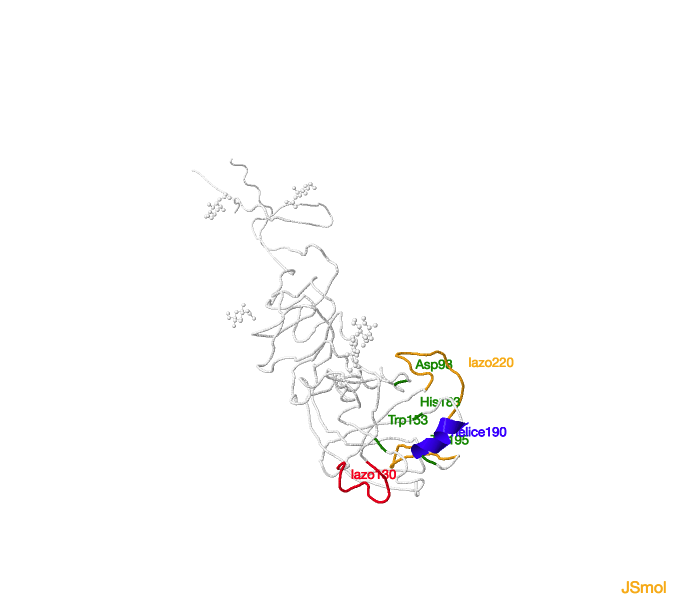
La región globular externa de la hemaglutinina es la región N-terminal, la subunidad HA1. Ésta contiene el bolsillo de unión a ácido siálico en el extremo de cada monómero. Este sitio de unión se caracteriza por
tener cuatro características estructurales en las distintas cepas virales: la
α-hélice 190 (en
azul en el modelo inferior), el
lazo 130 (en
rojo), el
lazo 220 (en
naranja) y una red de
aminoácidos conservados unidos por puentes de H: Asp98, Trp153, His183 y Tyr195, que constituyen el núcleo del bolsillo de unión al receptor en la subunidad HA1.
Bolsillo de unión a ácido siálico de la subunidad HA1.
Mecanismo de acción
En la conformación pre-fusión, ambas subunidades HA1 y HA2 están unidas por el puente disulfuro mencionado anteriormente (Figura 3). Es un enlace flexible cuya ruptura permitirá que HA adquiera una conformación que permite acercar
las dos membranas a fusionar.
El proceso de fusión de membranas se induce con una bajada de pH que ocurre en el endosoma después de la internalización del virus por endocitosis. La subunidad HA2 sufre un cambio conformacional inducido por el pH ácido
desde su conformación prefusogénica hasta que adquiere una conformación final en horquilla (Figura 1). Adicionalmente, se produce la apertura de las zonas globulares.
En el modelo siguiente se observa la conformación prefusogénica de la proteína HA, con la subunidad
HA2 (en
verde) y la
HA1 (en
naranja). El
péptido de fusión está señalado en
azul y se observa que queda rodeado por átomos tanto de la HA2 como de la HA1, es decir, no está expuesto. Para mayor claridad, solo se muestra el péptido de fusión correspondiente a un único monómero.
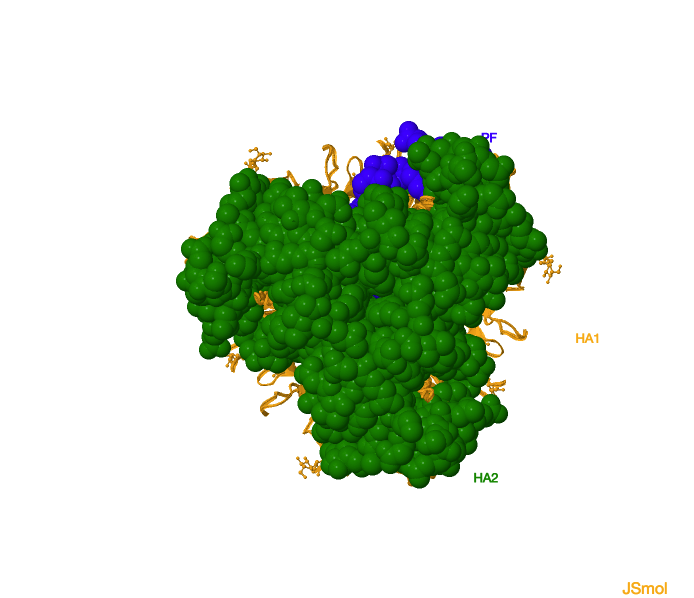
Conformación prefusogénica de la hemaglutinina. PF, péptido de fusión.
A pH ácido, la estructura prefusogénica sufre un cambio conformacional que determina la exposición el péptido de fusión de la subunidad HA2, y su inserción en la membrana del lisosoma, adquiriéndose la estructura en prehorquilla
(Figura 1 y modelo inferior).
Conformación postfusogénica de la HA2.
Posteriormente, un redoblamiento de la proteína determina que interaccionen las regiones de HA2 HR2 (en su extremo C-terminal próxima al dominio transmembrana) y HR1, próxima al péptido de fusión (Figura 3). Se aproximan
entonces la membrana diana (membrana del lisosoma) y la membrana del virus, produciéndose la fusión de ambas y la consecuente liberación de la nucleocápsida al interior celular. La proteína adquiere el estado final postfusión u horquilla (Figura 1).
En el siguiente enlace se muestra un vídeo resumen del mecanismo de acción de la proteína HA del virus de la gripe.
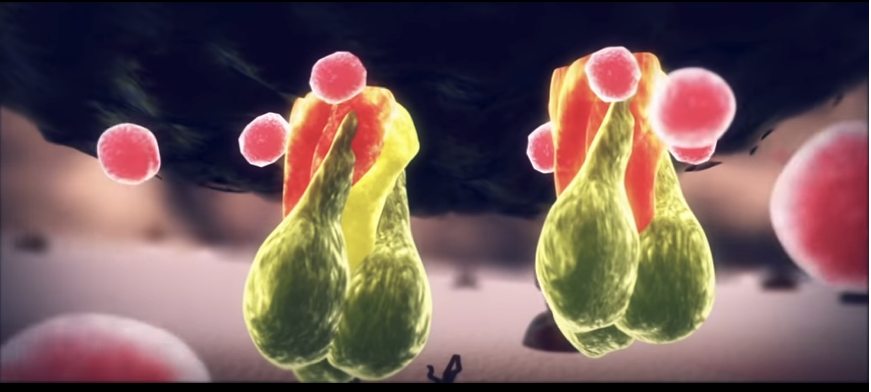 Vídeo animación del mecanismo de infección del virus de la gripe.
Vídeo animación del mecanismo de infección del virus de la gripe.
La proteína de fusión del virus del SIDA VIH
El genoma del Virus de la Inmunodeficiencia Humana (VIH) codifica para una única glicoproteína de la envuelta (Env), gp160. Durante su transporte hacia la superficie del virión, la intensa glicosilación que sufre gp160 provoca
su escisión en dos polipéptidos: gp120 y gp41.
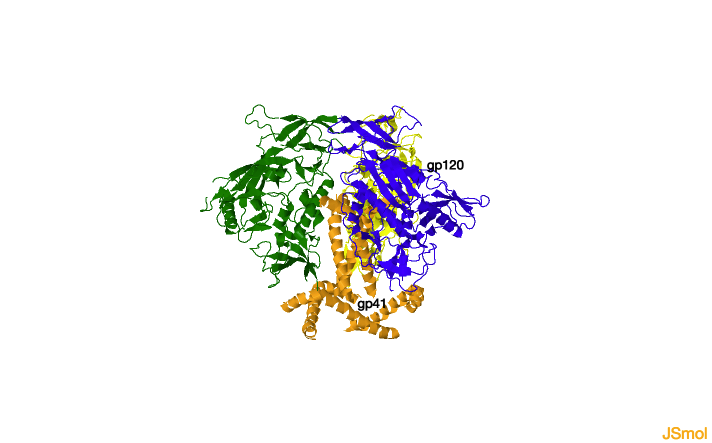
Env es una glicoproteína compuesta por tres monómeros idénticos. Cada monómero consiste en dos subunidades unidas no covalentemente: la subunidad N-terminal, gp120, localizada en la zona más externa del virión cuya función
es la unión al receptor, y la subunidad transmembrana,
gp41, que dirige la fusión de membranas. En el modelo inferior se representan la subunidad
gp41 en
naranja y las tres las tres subunidades gp120 en
amarillo,
azul y
verde.
Estructura de la proteína Env del VIH.
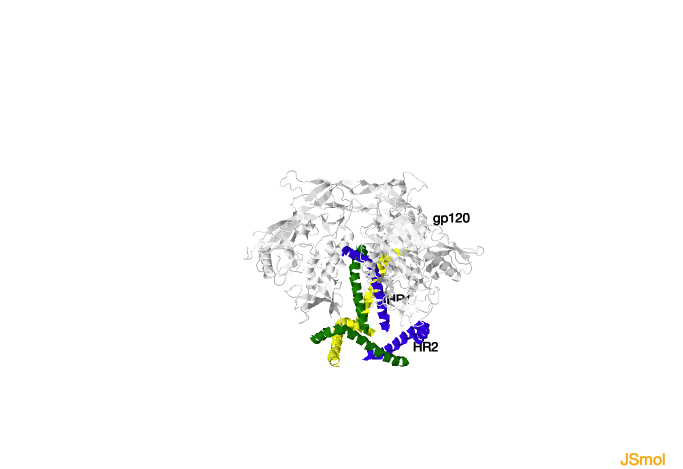
Las tres subunidades de gp41 constituyen un tronco que atraviesa la membrana vírica. Cada monómero contiene regiones conservadas entre diferentes proteínas de fusión víricas comentadas más arriba: en su extremo N-terminal el péptido de fusión;
las secuencias heptadas repetidas HR1 y HR2; y el dominio transmembrana (Figura 4). Puede compararse este esquema con el de la proteína HA2 mostrado en la Figura 3.
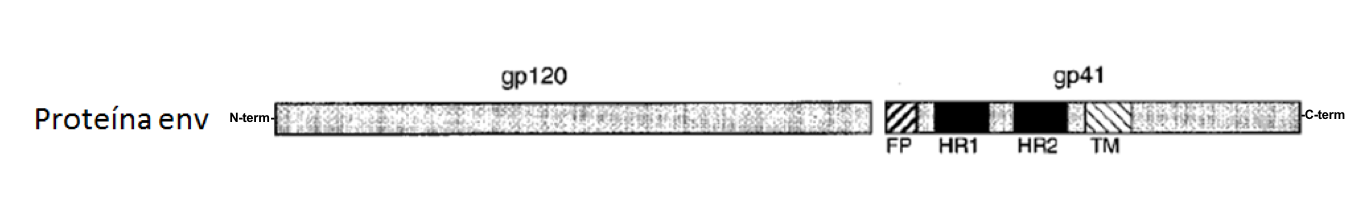 Figura 4. Esquema de la proteína Env (FP, péptido de fusión; HR, heptada repetida; TM, dominio transmembrana).
Figura 4. Esquema de la proteína Env (FP, péptido de fusión; HR, heptada repetida; TM, dominio transmembrana).
En el siguiente enlace se resaltan las regiones HR1 y HR2 de las tres subunidades de gp41, representadas con los colores
amarillo,
azul y
verde en su conformación prefusogénica.
Conformación prefusogénica del trímero env.
Mecanismo de acción
El primer paso en la infección vírica consiste en el reconocimiento de la célula diana. En el caso del VIH, la gp120 se une selectivamente al receptor CD4, un miembro de la familia de las inmunoglobulinas que se expresa en la
superficie de linfocitos T, linfocitos B, monocitos y otras células. CD4 es una glicoproteína transmembrana cuyo sitio de unión con Env implica una región de unos 30 residuos. La unión gp120-CD4 produce cambios conformacionales en
gp120, provocando una reorganización de las regiones hipervariables (V1-V5) que determina la exposición del sitio de unión a receptores de quimiocinas de la superficie celular: CCR5 y CXCR4, entre otros, que actúan como
correceptores (Figura 5).
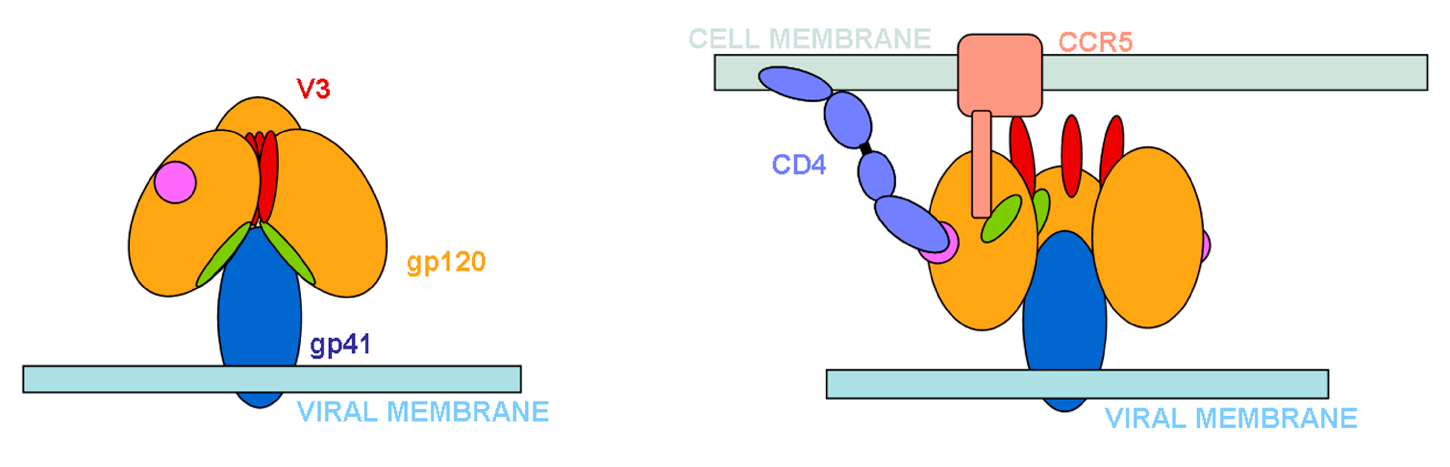 Figura 5: Unión de gp120 a CD4 y al receptor de quimiocinas CCR5. Tomado de Zanetti et al., 2006.
Figura 5: Unión de gp120 a CD4 y al receptor de quimiocinas CCR5. Tomado de Zanetti et al., 2006.
A su vez, la formación de complejos gp120-CD4-correceptor provoca cambios conformacionales en gp41, que expone el péptido de fusión e implican también a las regiones HR1 y HR2, dando lugar a la conformación pre-horquilla (intermediario).
Finalmente, ese intermediario deriva en una estructura final en horquilla helicoidal, en la cual los péptidos de fusión y los dominios transmembrana se colocan en el mismo extremo, en un mecanismo similar al descrito para el
virus de la gripe (ver Figura 1). Este cambio conformacional desencadena la fusión de membranas.
En el modelo inferior se puede apreciar la estructura postfusogénica de la gp41, donde las
hélices HR1 forman una estructura de tipo coiled-coil paralela (tres hélices empaquetadas en el interior) que queda rodeada por las tres
hélices HR2 del complejo en disposición antiparalela. Esta estructura postfusogénica, que es muy estable, se asemeja a la estructura postfusogénica de la proteína HA2 del virus de la gripe descrita más arriba.
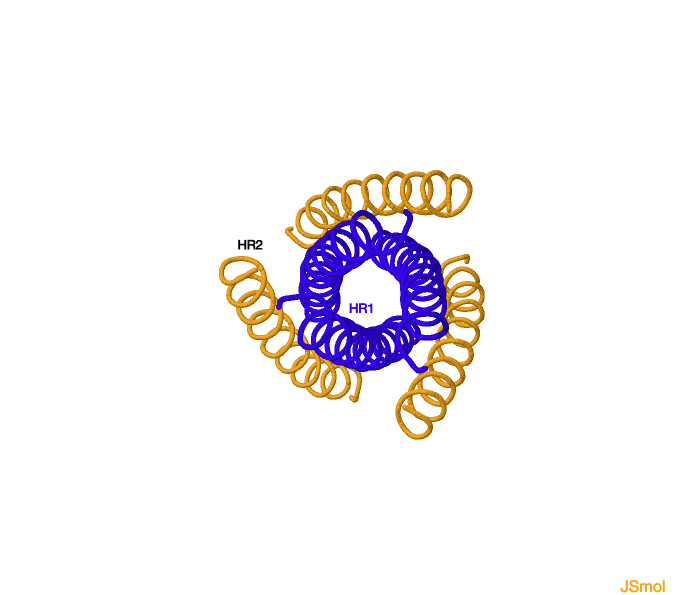
Conformación postfusogéniga de gp41.
En resumen, una vez que gp120 reconoce y une al receptor y correceptores, induce un cambio conformacional en gp41 que expone su péptido de fusión y desencadena la fusión de membranas. Aquí os dejamos un video con una animación del mecanismo de fusión de membranas inducida por la proteína gp41.
 Video resumen del mecanismo de fusión de membranas inducida por la proteína gp41 del VIH, y del mecanismo de acción de la Enfuvirtida (T-20).
Video resumen del mecanismo de fusión de membranas inducida por la proteína gp41 del VIH, y del mecanismo de acción de la Enfuvirtida (T-20).
Fármacos antivirales anti-fusión.
Existen ciertos ligandos que pueden retardar o bloquear la infección vírica al unirse selectivamente a la proteína de fusión en el paso previo a la fusión de las dos bicapas. Por ejemplo, mediante este mecanismo determinados anticuerpos
combaten la infección vírica.
La Enfuvirtida (antes T-20) es el primer antirretroviral utilizado como inhibidor de fusión vírica en pacientes infectados por VIH-1. Es un péptido análogo de la región HR2 de gp41. Su mecanismo de acción consiste
en la unión competitiva a la región HR1 de gp41 para impedir los cambios conformacionales de gp41 descritos más arriba tras la unión del VIH-1 a los receptores celulares (interacción HR1-HR2 que da lugar a la estructura postfusogénica
en horquilla), impidiendo el acercamiento y posterior fusión entre las membranas vírica y celular. En la segunda parte del vídeo anterior se resume el mecanismo de acción de este fármaco.
El principal obstáculo al que se enfrenta este tipo de tratamientos es la aparición de resistencias. Por ejemplo, se pueden dar mutaciones en varias posiciones de la proteína Env, incluyendo residuos de gp120, que confieran
resistencia frente a la Enfuvirtida. Por otra parte, algunas de las mutaciones que se producen en HR1 y que reducen la afinidad por T-20, parece que retardan la fusión al incrementarse la afinidad por anticuerpos contra la región próxima
a la membrana de gp41(Ac que neutralizan virus).
Guion elaborado por Alicia Mediavilla Carrancio, Lucía Méndez García, Fátima Pollo Rodríguez y Helena Villanueva Riesco, alumnas de Química e Ingeniería de Proteínas, 3er curso del Grado en Biotecnología, curso 2017/18.
Cuestiones
Responder al cuestionario correspondiente en la página de Studium
Referencias
Byrd-Leotis, L., Cummings, R.D., & Steinhauer, D.A. (2017). The Interplay between the Host Receptor and Influenza Virus Hemagglutinin and Neuraminidase. International Journal of Molecular Sciences 18, 7, pp.1541. Disponible en:
< http://doi.org/10.3390/ijms18071541>.
Bottaro, E.G. (2007). Enfuvirtide: el primer paso de una nueva estrategia de tratamiento antirretroviral. Medicina (B. Aires) 67, 2, 95-205. Disponible en:
<
http://www.scielo.org.ar/scielo.php?script=sci_arttext&pid=S0025-76802007000200016&lng=es&nrm=iso>.
Goodsell D., RCSB PDB Molecule of the Month doi:10.2210/rcsb_pdb/mom_2006_4
Goodsell D., RCSB PDB Molecule of the Month doi:10.2210/rcsb_pdb/mom_2014_1
Influenza animation-flu virus mechanism. https://www.youtube.com/watch?v=YSgkoldBNkI
Harrison, S.C (2008). Viral membrane fusion. Nature Structural & Molecular Biology, 15, 7, 690–698. Disponible en:
< http://doi.org/10.1038/nsmb.1456>.
HIV entry. https://www.youtube.com/watch?v=51z2dwdOLUE
Lamb, R.A. & Jardetzky, T.S. (2007). Structural basis of viral invasion: lessons from paramyxovirus F. Current Opinion in Structural Biology 17, 4, 427–436. Disponible en:
<
http://doi.org/10.1016/j.sbi.2007.08.016>.
Melikyan, G.B. (2011). Membrane fusion mediated by human immunodeficiency virus envelope glycoprotein. Current Topics in Membranes 68, 81–106. Disponible en:
<
https://www.ncbi.nlm.nih.gov/pubmed/21771496>.
PDB www.rcsb.org, H.M. Berman, J. Westbrook, Z. Feng, G. Gilliland, T.N. Bhat, H.
Weissig, I.N. Shindyalov, P.E. & Bourne (2000). The Protein Data Bank URL: Nucleic Acids Research, 28: 235-242.
Santana, A.; Domínguez, C.; Lemes, A.;Molero, T & Salido, E. (2003). Biología celular y molecular del virus de inmunodeficiencia humana (VIH). Rev Diagn Biol 52, 1, 07-18. Disponible en:
<
http://scielo.isciii.es/scielo.php?script=sci_arttext&pid=S0034-79732003000100001&lng=es&nrm=iso>.
Zanetti, G.; Briggs, J.A.G.; Grünewald, K.; Sattentau , Q.J. & Fuller, S.D. (2006). Cryo-Electron Tomographic Structure of an Immunodeficiency Virus Envelope Complex In Situ. PLOS Pathogens 2(8): e83.
Disponible en https://doi.org/10.1371/journal.ppat.0020083.