Introducción
Las caspasas son enzimas que juegan un rol importante en el mantenimiento de la homeostasis, puesto que catalizan la muerte celular programada o apoptosis. Actualmente en humanos se conocen unas 14 caspasas diferentes.
El nombre ‘Caspasa’,
Cisteín-
ASPártico prote
ASAS, se debe a su actividad como proteasa con una cisteína en su sitio catalítico, que ataca residuos que se encuentran después de un aspártico (en su extremo C- term), actuando a nivel del enlace peptídico.
Son proteínas muy conservadas evolutivamente.
La apoptosis es un proceso necesario para eliminar células que ya no deberían formar parte del organismo. Algunas células tienen que eliminarse durante el desarrollo embrionario (Figura 1); pero otras son anormales y pueden causar problemas en el resto
del organismo, por eso es importante que el proceso de muerte celular se desarrolle correctamente.
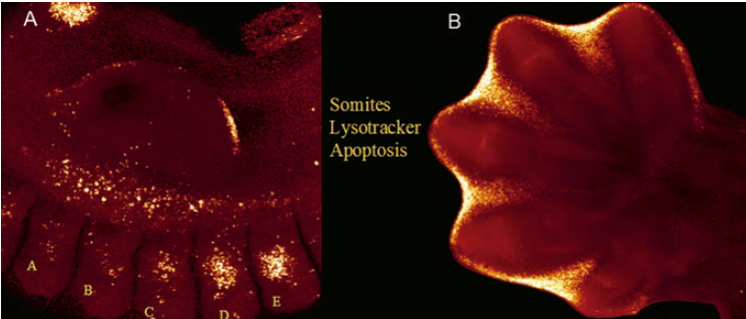
Figura 1. Apoptosis durante el desarrollo embrionario: los puntos brillantes muestran células en proceso de muerte celular programada en las regiones interdigitales durante el desarrollo embrionario de las extremidades de rata. De Zucker & Rogers, 2019.
En algunos casos, cuando una célula tiene el ADN dañado y no se puede reparar ni realizar apoptosis, se desarrolla un cáncer. A veces, las células inmunes lo detectan e intentan inducir la apoptosis por una vía externa. Así, las caspasas participan también
en la supresión de tumores (caspasas 2, 8 y 14). A parte de participar en la apoptosis y en la supresión tumoral, las caspasas están implicadas en procesos de inflamación (caspasas 1 y 11), y en la diferenciación y regeneración de
tejidos (caspasa 3).
Tipos de caspasas
Las caspasas humanas se pueden dividir en 3 grupos, basados en la similaridad de sus secuencias y en su función biológica:
- Caspasas inflamatorias: Son las caspasas 1, 4, 5, 11, 12, 13 y 14. De este grupo se ha caracterizado en profundidad la estructura de la caspasa 1. Su función es principalmente la piroptosis, una forma de muerte
celular programada que ocurre ante la infección por parte de un patógeno intracelular.
- Caspasas implicadas en apoptosis: Encontramos dos tipos:
- Iniciadoras. Son las caspasas 2, 8, 9 y 10. Su función consiste en la iniciación de la señal de apoptosis.
- Efectoras. Son las caspasas 3, 6 y 7. Su función consiste en una proteólisis masiva que resulte en apoptosis.
Estructura
Las caspasas pueden ser un peligro para la célula y por eso se sintetizan como proenzimas inactivas. Típicamente tienen una estructura que consiste en 3 partes:
- Un prodominio en el extremo N terminal
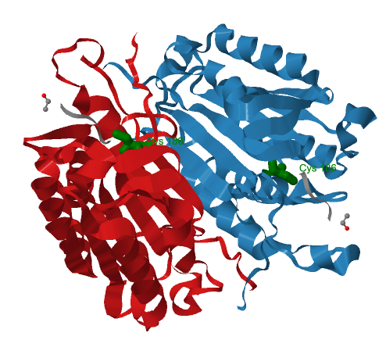
- Una
subunidad grande (p20, en rojo en el modelo siguiente) con cisteína en el centro activo, motivo muy conservado.
- Una
pequeña subunidad C-terminal (p10, en verde en el modelo).
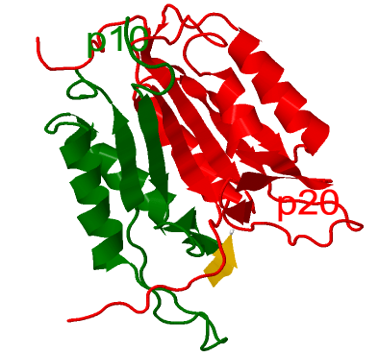 Heterodímero p20/p10 de las caspasas
Heterodímero p20/p10 de las caspasas
La longitud del pro-dominio depende del tipo de caspasa y puede tener más de 100 aminoácidos. El prodominio está dividido en dos subdominios: el ‘death effector domain’ o DED y el ‘caspase recruitment domain feature’ o
CARD. Este último consta de 6 ó 7 hélices alfa antiparalelas con un núcleo hidrofóbico (en rojo en el modelo siguiente) y residuos cargados en la superficie. El cofactor Apaf-1 también tiene un dominio CARD (en negro en el modelo) que interacciona con el dominio CARD de la caspasa 9 para formar luego el apoptosoma, explicado más abajo.
 Dominios CARD de la caspasa 9 interaccionando con dominios CARD de A paf-1
Dominios CARD de la caspasa 9 interaccionando con dominios CARD de A paf-1
El dominio
CARD se encuentra en las caspasas 1, 2, 4, 5, 9, 12 y 13. Por otro lado, el dominio DED se encuentra en las procaspasas 8 y 10. Ambos dominios reclutan caspasas iniciadoras hacia los complejos de inducción de muerte
o inflamación.
El prodominio se escinde de la subunidad grande en la secuencia AspX. Después de escindirse, p20 y p10 se organizan en un heterodímero donde pueden interaccionar. La asociación de dos heterodímeros, que interaccionan mayoritariamente
mediante sus regiones p10, forma el tetrámero proteolítico donde las unidades grandes encierran las pequeñas. La estructura puede verse en el modelo siguiente, con las
dos subunidades p10 interaccionado marcadas en verde.
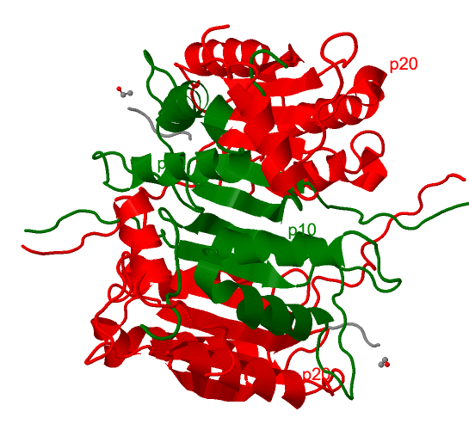 Los dos heterodímeros de la caspasa
Los dos heterodímeros de la caspasa
El sitio activo
Cada caspasa tiene dos sitios activos que funcionan generalmente de forma independiente. El sitio activo parece una cavidad formada por aminoácidos de ambas subunidades p20 y p10. Está bien definido y se encuentra en un
surco en la superficie de la proteína, según puede verse en el siguiente modelo en el que el sustrato se muestra en estilo esqueleto o “wireframe”.
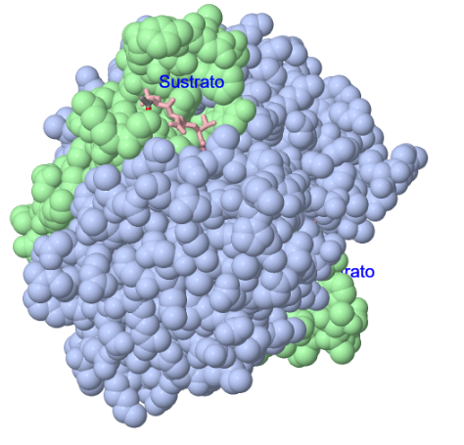 Los dos sitios activos de la caspasa 3
Los dos sitios activos de la caspasa 3
El sitio activo consiste en 4 lazos superficiales (L1, L2, L3 y L4) del mismo monómero (ver Figura 2). L1 y L4 se encuentran a ambos lados del surco de unión al sustrato y L3 forma la base. L2 se encuentra sobre el surco y contiene
el
residuo catalítico cisteína 186 (en
verde en el modelo mostrado más abajo).
 Caspasa 7 con las cisteínas del centro activo del lazo 2 marcadas en verde
Caspasa 7 con las cisteínas del centro activo del lazo 2 marcadas en verde
En la conformación activa, gira 90° liberando la cisteína para la unión del substrato (Figura 2A). L2’ del otro monómero estabiliza la “conformación activa” del sitio activo por interacciones con L2 y L4. Por lo tanto, en conformación inactiva L2’ está en
conformación cerrada (ver figura 2B) y gira 180° para adquirir su conformación
activa estabilizando L4 y L2.
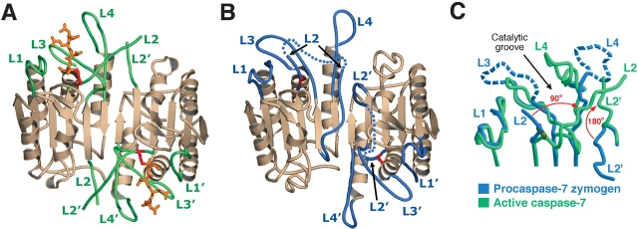 Figura 2.mecanismo molecular de la activación de la caspasa 7.
Figura 2.mecanismo molecular de la activación de la caspasa 7.
A.Caspasa 7 activada y unida al susstrato (en naranja). La Cys del sitio activo está maracada en rojo. B.Procaspasa 7 con L2 en conformación cerrada. C.Comparación de la procaspasa 7 (azul) y caspasa 7 activa (verde). Tomado de Shi, 2004.
El sitio activo se divide en cuatro subsitios, S1, S2, S3 y S4 y reconoce una secuencia corta del sustrato de 4 aminoácidos P1-P4 (Pn-P4-P3-P2-P1/P1´-P2´-Pn´) (/, sitio de corte del sustrato), que se unen respectivamente a los diferentes subsitios, siendo
la secuencia más frecuente DXEX(P1-P4). Las diferencias en la especificidad del subsitio S4 por P4 determinan la clasificación de las caspasas en tres grupos, grupo I (P4 residuo hidrofóbico, caspasas 1,
4, 5, 13), grupo II (P4 Asp, caspasas 2, 3, 7) y grupo III (P4 residuo alifático, caspasas 6, 8, 9, 10).
La unión del sustrato a la caspasa está estabilizada por puentes de H entre
Arg179,
Gln283
y
Arg341 (que están conservados en todas las caspasas) y el residuo P1 del sustrato (Asp). El ensamblaje de S1 es muy restrictivo y por eso solo soporta Asp en P1.
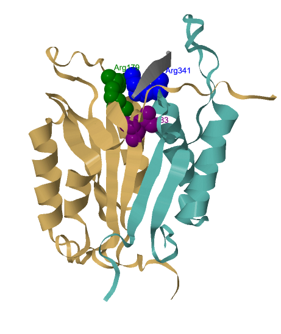 Estructura de la caspasa 3 mostrando los residuos Arg 179, Gen 283 y Arg 341 que estabilizan la unión con el sustrato en S1.
Estructura de la caspasa 3 mostrando los residuos Arg 179, Gen 283 y Arg 341 que estabilizan la unión con el sustrato en S1.
Los subsitios de unión S2 y S3 son menos restrictivos y aceptan diferentes residuos en P2 y P3 respectivamente. Los bolsillos S2 de las caspasas 3, 6 y 7 son más pequeños que los de otras caspasas, por lo que se unen a residuos
P2 más pequeños como Ala y Val. El sitio S3 presenta una Arg conservada en todas las caspasas, lo que sugiere que Glu será el residuo preferido en posición P3 para interactuar con este subsitio. También la Ser339 está conservada
en la mayoría de las caspasas. En el siguiente modelo se resalta la interacción de la
Ser339 (en verde) y
Arg341 (en azul cielo) con el sustrato, un tetrapéptido.
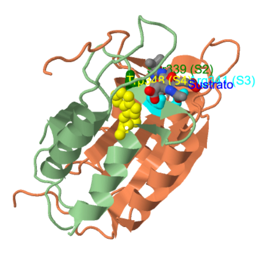 El sustrato estabiliza su unión a la caspasa 3 mediante puentes de H con Ser 339 y Arg 341 en S2 y S3; el Trp 348 contribuye a la geometría de S4.
El sustrato estabiliza su unión a la caspasa 3 mediante puentes de H con Ser 339 y Arg 341 en S2 y S3; el Trp 348 contribuye a la geometría de S4.
Como ya se ha comentado, el sitio de unión S4 determina la especificidad de unión con el sustrato (unión al residuo P4), por lo que la geometría y química de este subsitio varía entre las distintas familias de las caspasas.
La geometría de S4 viene determinada en parte por el
Trp348 conservado en las caspasas II y III pero no I (en amarillo en el modelo anterior) y por un lazo de superficie que se encuentra encima del sitio activo.
Activación
Podemos considerar de manera independiente la activación de las caspasas, y la activación de la cascada de apoptosis. Respecto a la activación de las caspasas, éstas están reguladas post-transcripcionalmente, puesto que
todas son sintetizadas como zimógenos (precursor enzimático inactivo) o dímeros inactivos. La activación empieza con la dimerización, seguida por una escisión intracatenaria o cleaving, proceso mediado por las caspasas iniciadoras.
La escisión aumenta de manera importante la actividad catalítica al producir cambios conformacionales del sitio activo. Como se ha comentado más arriba, la enzima madura es un heterotetrámero formado por dos heterodímeros p20/p10,
con dos centros activos.
La diferencia de activación entre caspasas efectoras e iniciadoras es que las efectoras son activadas proteolíticamente por otras caspasas y las iniciadoras gracias a interacciones proteína-proteína. Se conocen tres mecanismos de activación de estas enzimas:
- Activación por otra caspasa: Es una estrategia muy utilizada en la activación de caspasas efectoras (pro-dominio corto). Consiste en la activación de una caspasa por otra que ya está activada. La caspasa activa corta
el pro-dominio de la subunidad p20 y también separa la subunidad p20 de la subunidad p10 gracias a la presencia de aspartato en esta posición.
- Activación inducida por proximidad: Los receptores de muerte celular se agregan y forman un complejo de señalización en la membrana que atrae a varias moléculas de procaspasa 8, la caspasa iniciadora principal.
La actividad proteásica de la procaspasa 8 a pesar de ser baja es suficiente para permitir que las proenzimas se corten mutuamente activándose unas a otras debido a la alta concentración de zimógeno.
- Activación por asociación con una subunidad reguladora: El procesamiento proteolítico de la caspasa 9 tiene un bajo efecto en su activación. Para conseguir activar la enzima es necesaria la asociación con un cofactor,
Apaf1
(en azul en el modelo siguiente) y con el
citocromo c (en rojo) liberado por la mitocondria. El cofactor Apaf1 y el citocromo c se asocian en un proceso dependiente de ATP y atraen a procaspasas 9 formando el apoptosoma. Así, la activación se debe a un cambio
conformacional de la caspasa y no a una proteólisis.
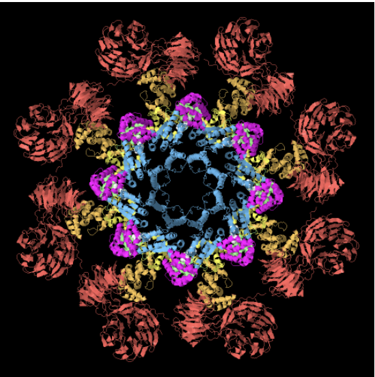
 Modelo del apoptosoma humano, mosttando las siete subunidades de Apaf1 en azul y el citocromo C en rojo.
Modelo del apoptosoma humano, mosttando las siete subunidades de Apaf1 en azul y el citocromo C en rojo.
En el siguiente modelo se observa la estructura del apoptosoma de D. melanogaster:
En cuanto a la apoptosis, existen dos vías de activación o cascadas de señalización: la extrínseca y la intrínseca, resumidas en el esquema de la Figura 3:
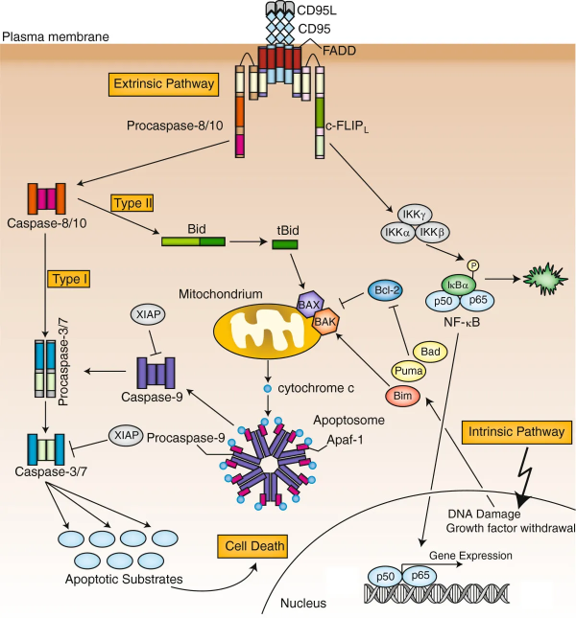 Figura 3. Esquema de las vías extrínseca e intrínseca de activación de la apoptosis tomado de Schleich & La Vrile, 2013.
Figura 3. Esquema de las vías extrínseca e intrínseca de activación de la apoptosis tomado de Schleich & La Vrile, 2013.
La vía extrínseca de apoptosis se induce por estímulos extracelulares o
ligandos que se unen a receptores transmembrana de muerte celular como CD95 o TRAIL-R (Figura 3), cuyo dominio extracelular es rico en cisteína y presentan un dominio citoplasmático responsable de la apoptosis. Pueden ser de
varios tipos:
- Receptores del factor de necrosis tumoral (TNF)
- Receptores Fas, con un dominio intracelular que al unirse a su ligando Fas activa las caspasas iniciadoras 8 y 10, que a su vez activan las caspasas efectoras 3 y 7, y/o la proteína Bid de la familia Bcl-2 que conducen
a la activación de endonucleasas. Este tipo de receptores se encuentran en células natural killer o en células infectadas por virus.
La vía intrínseca se desencadena por estímulos intracelulares como lesiones en el material genético, aumentos en la concentración del ion calcio o de ROS, que conducen a la síntesis de la proteína p53, que a su vez induce la síntesis de las proteínas Bax y Bak de la familia de BCL2, que aumentan la permeabilidad de la membrana mitocondrial externa de manera que se libera al citosol el citocromo c y los diferentes
componentes se unirán para formar el apoptosoma. Este complejo se une a la procaspasa 9, que deriva en la caspasa 9 activa, la cual inicia la cascada que desemboca en la activación de las caspasas efectoras 3 y 7. Estas
últimas se encargan de activar una enzima desoxirribonucleasa que conduce a la fragmentación del DNA y, por tanto la apoptosis.
Una vez se ha formado el apoptosoma, la decisión de muerte celular es irreversible e irrevocable, el proceso debe llevarse a cabo.
Mutaciones y enfermedades relacionadas con las caspasas
Dado el importante papel de las caspasas en la regulación de la muerte celular, es natural que alteraciones de diversa naturaleza en las mismas provoquen modificaciones del funcionamiento normal del organismo, pudiendo causar
diferentes enfermedades, como veremos a continuación.
El nombre ‘Caspasa’, Cisteín-ASPártico proteASAS, se debe a su actividad como proteasa con una cisteína en su sitio catalítico, que ataca residuos que se encuentran después de un aspártico (en su extremo C- term), actuando a nivel del enlace peptídico.
Son proteínas muy conservadas evolutivamente.
Alzheimer. En los cerebros post-mórtem de pacientes con alzheimer hay un aumento en la expresión de mRNA de múltiples caspasas en comparación con cerebros control. A nivel proteico hay un aumento en la concentración de las caspasas
2, 3, 6 y 9. La caspasa 2 puede inducir la muerte celular sin necesidad de caspasas efectoras; además se ha demostrado que tiene un papel clave en la neurodegeneración, por lo que esta caspasa podría ser una diana en el tratamiento
de Alzheimer.
Enfermedades metabólicas. En la obesidad se acumulan grandes cantidades de lípidos y se da una expansión del tejido adiposo blanco debido al aumento del número y tamaño de los adipocitos. El aumento en el tamaño del adipocito
lleva a su muerte por apoptosis producida por unos niveles elevados de caspasas 3, 7 y 9. Los macrófagos se agregan en torno al cuerpo apoptótico y los restos de lípidos. Se liberan factores inflamatorios que contribuyen a la inflamación
del hígado y la muerte de células hepáticas.
Cáncer. En muchos tipos de cáncer se encuentra la proteína p53 mutada de manera que no se induce apoptosis, volviéndose la célula inmortal. Por otra parte, el cáncer puede deberse a alteraciones
en los genes que codifican las caspasas, existiendo numerosos polimorfismos (Tabla 1). Se ha descubierto que en determinados cánceres se produce la fosforilación de algunos residuos durante la mitosis por la quinasa dependiente de
ciclina 1 como consecuencia de una mutación tipo SNV (o de un solo nucleótido). En consecuencia, la activación de la cascada apoptótica por proteólisis queda inhibida. Esto se ha observado, por ejemplo, en tejido mamario y en linfocitos,
así como en células NK, lo cual conduce a inmunodeficiencias. También se ha descrito una deleción de 6 nucleótidos en el promotor del gen CASP8 que, y pese a reducir considerablemente su expresión, parece reducir el riesgo de sufrir
cáncer de mama entre otros.
Tabla 1. Tabla de mutaciones que afectan a los distintos tipos de caspasas y a qué tipo de cáncer predisponen. (Tomado de Olsson & Zhivotovsky, 2011).
Caspasas
Mutación
Expresión de la proteína
Cáncer
CASP8
Metilación del promotor
Baja-ausente
Neuroblastoma infantil
CASP8
Desplazamiento del marco de lectura
NA
Hepatocelular
CASP10
NA*
Baja-ausente
Pulmón-mama
CASP6
NA
Baja-ausente
Gástrico
CASP7
Sin sentido
NA
Colorectal
CASP3
NA
Baja-ausente
De mama
* En la siguiente tabla se recoge el papel de distintas mutaciones en las diferentes caspasas y el tipo de cáncer con el que se relacionan.
Guion elaborado por Raquel Benéitez Martínez, Anneleen De Winter, Mar Fernández Monzón y Lena Wantiez, alumnas de Química e Ingeniería de Proteínas, 3er curso del Grado en Biotecnología, curso 2019/20.
Cuestiones
Responder al cuestionario correspondiente en la página de Studium
Referencias
Bulotta, A., Farilla, L., Hui, H. & Perfetti, R. (2004). The role of GLP-1 in the regulation of islet cell mass. Cell Biochem Biophys. 40:65-78.
Gene Cards, CASP 8 gene, https://www.genecards.org/cgi-bin/carddisp.pl?gene=CASP8
Goodsell D. RCSB PDB 101, Molecule of the month. Apoptosome. doi:10.2210/rcsb_pdb/mom_2014_9
Goodsell D. RCSB PDB 101, Molecule of the month. Caspases. doi:10.2210/rcsb_pdb/mom_2004_8
Julien, O. & Wells, J.A. (2017). Caspases and their substrates. Cell Death & Differentiation 24:380-1389.
Nicholson, D.W. (1999). Caspase structure, proteolytic substrates, and function during apoptotic cell death. Cell Death & Differentiation 6:1028-1042.
Olsson, M. & Zhivotovsky, B. (2011). Caspases and cancer. Cell Death & Differentiation 18:1441–1449. doi:10.1038/cdd.2011.30
PDB www.rcsb.org, H.M. Berman, J. Westbrook, Z. Feng, G. Gilliland, T.N. Bhat, H. Weissig, I.N. Shindyalov, P.E. Bourne (2000) The Protein Data Bank URL: Nucleic Acids Research, 28: 235-242.
Rotonda, J., Nicholson, D.W., Fazil, K.M., Gallant, M., Gareau, Y., Labelle, M., Peterson, E.P., Rasper, D.M., Ruel, R., Vaillancourt, J.P., Thornberry, N.A. & Becker, J.W. (1996). The three-dimensional structure of apopain/CPP32,
a key mediator of apoptosis. Nat Struct Biol. 3:619-625.
Schleich, K. & Lavrik, I.N. (2013). Mathematical modeling of apoptosis. Cell Communication and Signaling 11:44. http://www.biosignaling.com/content/11/1/44
Shalini, S., Dorstyn, L., Dawar, S., & Kumar, S. (2015). Old, new and emerging functions of caspases. Cell Death & Differentiation 22:526–539. doi:10.1038/cdd.2014.216
Shi, Y. (2004). Caspase activation, inhibition, and reactivation: a mechanistic view. Protein science 13:1979–1987. doi:10.1110/ps.04789804.
The UniProt Consortium. UniProt: a worldwide hub of protein knowledge. Nucleic Acids Res. 47: D506-515 (2019)
Troy, C.M. & Jean, Y.Y. (2015) Caspases: Therapeutic Targets in Neurologic Disease. Neurotherapeutics 12:42-48. doi: 10.1007/s13311-014-0307-9.
van Raam, B.J. & Guy S. Salvesen G.S. (2013). Caspase-8 and Caspase-10. En Handbook of Proteolytic Enzymes (Third Edition), Rawlings N.D. and Salvesen, G. Eds
Wilson C.H. & Kummar S. (2018). Caspases in metabolic disease and their therapeutic potential. Cell Death & Differentiation 25, 1010-1024. doi: 10.1038/s41418-018-0111-x.
Zucker, R.M. & Rogers J.M. (2019) Confocal Laser Scanning Microscopy of Morphology and Apoptosis in Organogenesis-Stage Mouse Embryos. Methods in Molecular Biology 1965:297-311. doi: 10.1007/978-1-4939-9182-2_20.