INTRODUCCIÓN
Retinoblastoma (Rb) fue el primer supresor tumoral descrito y, como sabemos hoy en día, mutaciones en su estructura condicionan una gran cantidad de cánceres.
Estructuralmente, hablamos de una proteína de gran tamaño y multifuncional; la cual se organiza en distintos dominios. La actividad de esta proteína se regula, principalmente, por fosforilación en múltiples sitios descritos a lo largo de los años y se
encuentra implicada en mecanismos de control del ciclo celular, apoptosis y senescencia.
ESTRUCTURA
Estructura General
La proteína RB tiene un total de 928 residuos y tres dominios diferenciados.
El dominio N-terminal (RBN) tiene estructura globular formada por dos lóbulos A y B, cada uno de los cuales tiene estructura de motivo de ciclina (motivo compuesto por 5 hélices alfa, hay tres centrales que forman una región hidrofóbica central del dominio,
y dos hélices adicionales que se empaquetan contra las primeras). Los dos lóbulos están separados por un núcleo hidrofóbico. Este dominio podría estar implicado en las interacciones proteína-proteína.
El dominio C-terminal (RBC) carece de una estructura ordenada, pero es necesario para la unión de E2F-DP.
El dominio central, pRbAB se conoce con el nombre de bolsillo. Es la región mínima necesaria para la unión de oncoproteínas. Contiene dos cadenas, A y B.
Las dos subunidades, denominadas cadenas A y B, son distintas entre sí, aunque en las estructuras secundarias de ambas predominan las hélices alfa.
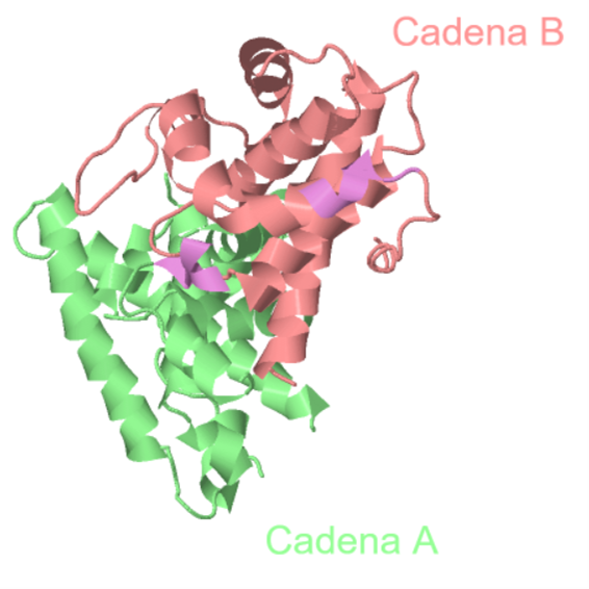 Dominio central de Rb – Cadenas A y B (remarcadas las hélices 310)
Dominio central de Rb – Cadenas A y B (remarcadas las hélices 310)
Cadena A
La cadena A presenta diez hélices ɑ de longitudes variables (6-20 aminoácidos) separadas por giros proteicos. Destaca también una hélice 310 en torno a Lys561.
Cadena B
La cadena B, por su parte, está formada por siete hélices ɑ, también separadas por giros. Existen dos hélices 310 en torno a Glu693 y Gln770. A diferencia de la cadena A, existen también dos hebras ß de corta longitud (3
aminoácidos), que conforman una horquilla beta. Entre ambas cadenas existe una interfaz hidrofóbica que las une.
FUNCIÓN
La pérdida de la función de la proteína del retinoblastoma, pRb, contribuye al desarrollo de la mayoría de las neoplasias. La pRb regula el ciclo celular, apoptosis y diferenciación. El proceso de actuación de la pRb viene determinado por la capacidad
que tiene de unir proteínas que actúan como factores de transcripción, evitando de esta manera su actividad.
La proteína RB tiene como función principal inhibir la transición de la fase G1 a S. Su actividad depende de su estado de fosforilación: si RB no está fosforilada (estado activo), se encuentra unida al factor de transcripción E2F evitando su translocación
al núcleo y la activación de genes necesarios para la síntesis del ADN; y si se fosforila, por complejos ciclina-CDK, el E2F se libera y se produce proliferación celular
Así los efectos inhibitorios de esta proteína se deben a la regulación que ejerce sobre la familia de factores de transcripción E2F.
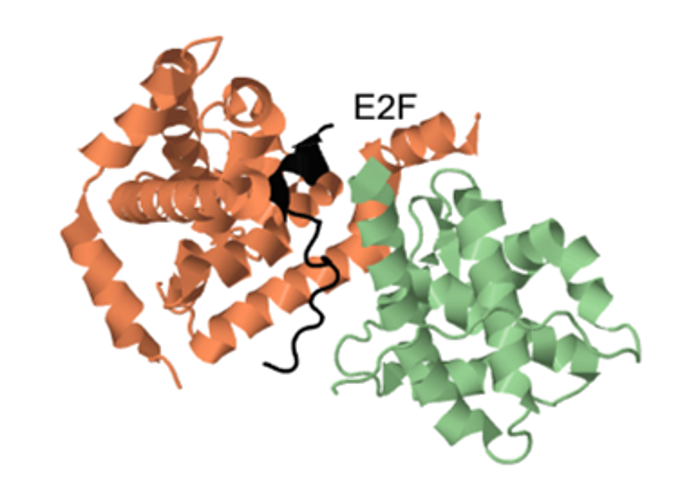 Interacción de Rb con E2F
Interacción de Rb con E2F
Relación con E2F y el ciclo celular
E2F es un factor de transcripción involucrado en el control del ciclo celular. Regula la expresión de genes necesarios para entrar en la fase S y el paso de su punto de control. Para unirse al ADN, los factores E2F necesitan
formar un dímero con una proteína DP (Dimerization Partner). Hay E2F activadores y represores de la transcripción, los cuales pertenecen una familia de factores conservados.
La unión de la proteína pRB a los E2F activadores, impide la transcripción de sus genes diana. La transcripción de la proteína Cdk2 y la ciclina E (necesarios para el progreso de G1 a S) también es estimulada por los factores E2F. Los complejos formados
por ciclina D y Cdk4 o Cdk6 tienen que fosforilar la proteína pRB para la liberación del E2F.
En el dominio Central pRbAB, en la interfase A-B existe un núcleo hidrofóbico, creado por las cadenas laterales de 20 residuos. A este bolsillo se une E2F a través de las interacciones mostradas enlace y en la tabla:
Zona de interacción de Rb con E2F

RELACIÓN ENTRE RB Y EL CÁNCER
El gen supresor de tumores que codifica retinoblastoma (RB) está inactivado funcionalmente en gran cantidad de cánceres. Sus funciones juegan un papel fundamental durante todo el proceso cancerígeno
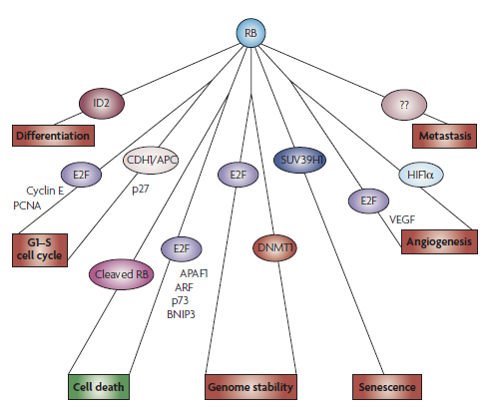 Relación entre la proteína retinoblastoma y factores relacionados con el cáncer.
Relación entre la proteína retinoblastoma y factores relacionados con el cáncer.
En la iniciación del cáncer
Re-entrada en el ciclo celular
RB promueve la diferenciación de múltiples tejidos y regula gran cantidad de factores de transcripción y complejos de remodelación de la cromatina implicados en el control del ciclo celular. RB no funcional puede causar la
entrada de nuevo en ciclo de una célula ya diferenciada, y esto es debido a que RB modula la estructura y localización de la heterocromatina en las células senescentes, de tal forma que, si esta proteína no actúa, los genes relacionados con
el ciclo celular podrán expresarse y dar lugar a cáncer.
Papel en la reparación del DNA
Daños en el DNA incrementan la acetilación de RB, de tal forma que disminuye la fosforilación de esta proteína volviéndola más activa y por tanto incrementando su represión sobre E2F1.
En la progresión del cáncer
< Pérdida de función e incremento de la autofagia en respuesta a la hipoxia
Durante la progresión tumoral, los tumores sólidos logran un tamaño en el que el entorno hipóxico limita el crecimiento. Recientes estudios han demostrado que la presencia de RB funcional puede prevenir esta autofagia en respuesta
a la hipoxia mediante la inhibición de los genes diana de E2F. De tal forma que facilita la supervivencia de tumores tempranos.
Pérdida de función e inestabilidad cromosómica
Los tumores avanzados presentan un alto grado de inestabilidad genómica. RB podría ayudar a mantener la estabilidad genómica mediante la asociación a complejos reguladores de la cromatina, regulando así la estructura de la
cromatina (especialmente la región de los centrómeros y los telómeros) y por tanto la expresión génica. Muchos de estos complejos se asocian a RB por su dominio LXCXE, y se ha demostrado que mutaciones en este dominio contribuyen a
la inestabilidad y segregación errónea de los cromosomas debido a fallos en la formación de la heterocromatina pericéntrica.
La pérdida de RB impide la inducción de la senescencia celular.
La actividad del complejo RB-E2F y así como la interacción entre RB y enzimas remodeladoras de la cromatina como SUV39H1 median en la senescencia, actuando directamente en los loci de los genes del ciclo celular. Además, la
familia RB controla la longitud de los telómeros y media en las señales celulares para inducir senescencia en células con telómeros acortados. De tal forma que la pérdida de función de RB permite a las células tumorales evitar la detención
de su ciclo celular asociada al fenómeno de senescencia y permitir la progresión del cáncer.
Pérdida de función y promoción de la angiogénesis.
Algunas de las sustancias secretadas por las células cancerígenas para la formación de nuevos vasos, tales como VEGF (factor de crecimientos endotelial vascular) son reprimidas por la vía RB-EF
ONCOPROTEÍNA E7 Y RETINOBLASTOMA
El virus del papiloma humano (VPH) codifica diversas proteínas entre las que se encuentra E7. Esta molécula es capaz de interaccionar con un gran número de proteínas, algunas de las cuales participan en la regulación del ciclo celular como puede ser retinoblastoma.
A través de las interacciones E7 induce la progresión del ciclo celular y la inhibición de la diferenciación celular. Todas estas interacciones de E7 contribuyen al desarrollo de algunos tipos de cánceres como el cáncer de cuello uterino.
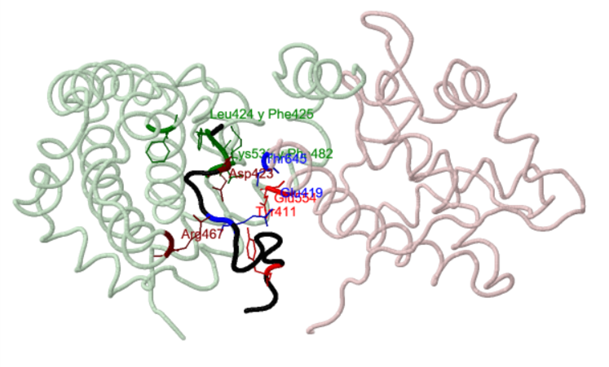
La oncoproteína E7 del virus del papiloma humano consta de nueve residuos con homología LxCxE que se unen al surco de la caja B de RB, de tal forma que impiden la unión de retinoblastoma a E2F. Las cadenas laterales de Leu 22, Cys 24, Glu 26 y Leu 28
alternadas se introducen en la ranura de la caja B y establecen uniones intermoleculares, mientras que los residuos restantes (Asp 21, Tyr 23, Tyr 25, Gln 27 y Asn 29) no participa directamente en la unión. En los extremos, la leucina
22 y la leucina 28, y en el centro, las cadenas laterales Cys 24 del péptido crean interacciones hidrofóbicas en el interior del surco. Los enlaces de hidrógeno se establecen por los grupos carbonilo de los residuos 22, 23 y 24, por
el grupo amida del residuo 24 y por el grupo carboxilo Glu 26 del péptido. La formación compleja entierra un total de 1.150 Å2 de superficie.
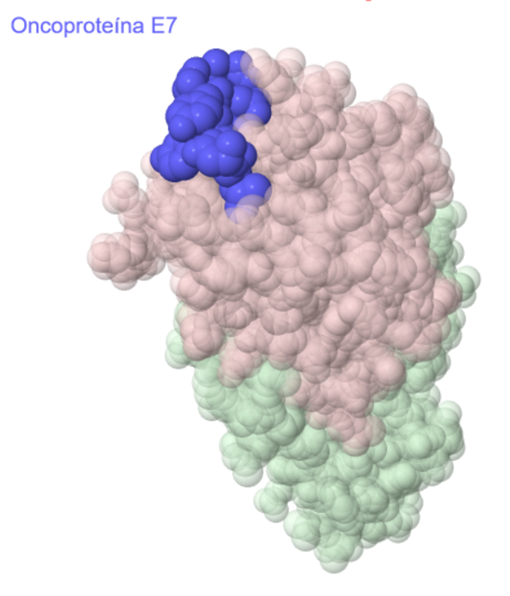 Unión de Rb a un péptido derivado de la oncoproteína E7
Unión de Rb a un péptido derivado de la oncoproteína E7
 Detalle del motivo LxCxE de la oncoproteína E7 en su unión al surco de la caja B de RB
Detalle del motivo LxCxE de la oncoproteína E7 en su unión al surco de la caja B de RB
Autores
Guion elaborado por Enrique Álvarez, Jordi Asís, Miguel Bastos y Víctor Mayo, alumnos de Química e Ingeniería de Proteínas, 3er curso del Grado en Biotecnología, curso 2019/20.
BIBLIOGRAFÍA
► Bonelli, M., La Monica, S., Fumarola, C., & Alfieri, R. (2019). Multiple effects of CDK4/6 inhibition in cancer: from cell cycle arrest to immunomodulation. Biochemical pharmacology, 113676.
► Henley, S.A., Dick, F.A. The retinoblastoma family of proteins and their regulatory functions in the mammalian cell division cycle. Cell Div 7, 10 (2012)
► Classon, M., & Dyson, N. (2001). p107 and p130: versatile proteins with interesting pockets. Experimental cell research, 264(1), 135-147.
► Kent, L.N., Leone, G. The broken cycle: E2F dysfunction in cancer. Nat Rev Cancer 19, 326–338 (2019)
► López Marure, R. LA REGULACIÓN DEL CICLO CELULAR Y EL CÁNCER. Vertientes. Revista Especializada en Ciencias de la Salud, 6(1).
► Burkhart, D., Sage, J. Cellular mechanisms of tumour suppression by the retinoblastoma gene. Nat Rev Cancer 8, 671–682 (2008)
► Le, J.-O., Russo, A. A. & Pavletich, N. P. Structure of the retinoblastoma tumor-suppressor pocket domain bound to a peptide from HPV E7. Nature 391, 859-865 (1998)