INTRODUCCIÓN
Las DNA metiltransferasas son proteínas enzimáticas que intervienen en la epigenética de los organismos. Son las encargadas de metilar zonas del DNA para controlar su función. La metilación generalmente se emplea para inhibir la transcripción
génica, es decir, cuando se metilan nucleótidos de determinados genes se impide la accesibilidad de la DNA polimerasa y, por tanto, la generación de RNA.
Este proceso es fundamental en el desarrollo embrionario, por el fenómeno de impronta genética. Este proceso consiste en que la expresión de ciertos genes se produce específicamente, dependiendo del sexo del progenitor. En el
estadio embrionario, normalmente ocurre una desmetilación del DNA y posteriormente ocurre una metilación de novo, dependiente de los distintos tejidos del organismo. Dos de los cuatro nucleótidos, adenina (A) y citosina (C), son susceptibles
de sufrir metilaciones. Esto se lleva a cabo de la siguiente manera:
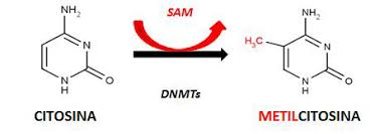
Existen distintas metiltransferasas, entre las que destacamos:
1. DNMT3: es la enzima encargada de metilar zonas del genoma durante los estadios iniciales de la vida, en el óvulo fecundado. Es decir, esta enzima se encarga de metilar de novo, zonas que no estaban metiladas con anterioridad.
2. DNMT1: mantiene la metilación durante la vida del organismo, lo que se llama metilación de mantenimiento. Cuando el DNA se replica, se metilan las regiones necesarias del genoma. La mayor parte de los grupos metilos se suelen
añadir sobre citosinas seguidas de guaninas (secuencias del tipo ----CG-----, -------GC-------)
3. DNA metiltransferasa de bacterias (HhaI): la metilación del material genético en estos organismos es importante ya que distingue su DNA del exógeno. Es decir, si un virus infecta una bacteria, enzimas de restricción pueden
eliminar a este parásito ya que su DNA no se encuentra metilado y se distingue de esta forma del de la bacteria.
A continuación, veremos con más detalle cada una de las enzimas.
1. DNMT3:
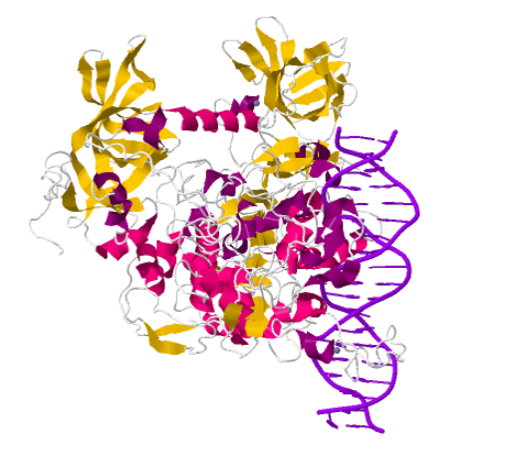
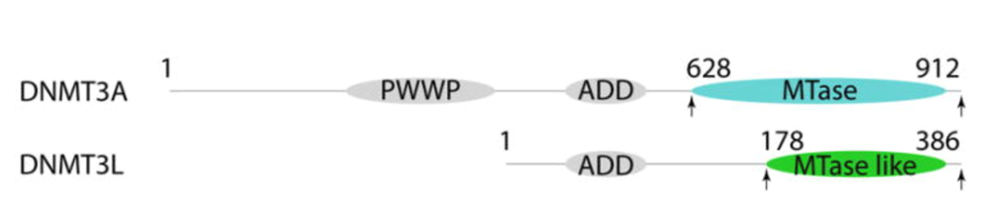
ESTRUCTURA GLOBAL
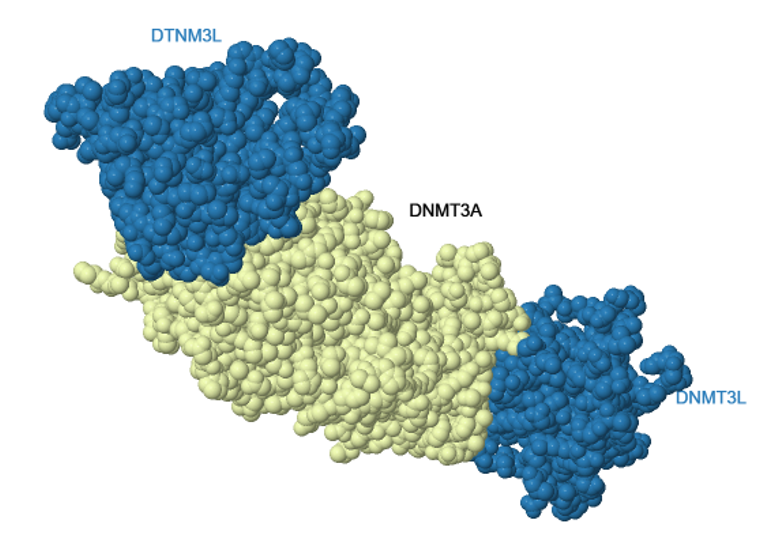
Se han descrito tres tipos de DNMT3: A, B y L. Las dos primeras se encargan de la metilación de novo del DNA, mientras que la tercera regula la actividad de las dos anteriores. De hecho, se ha demostrado que DNMT3L es indispensable
para la correcta metilación del DNA en estadios iniciales del desarrollo.
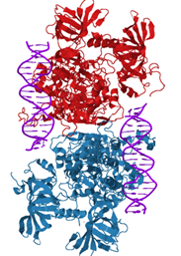
En nuestro modelo, tenemos obtenidas por cristalografía las proteínas 3A y 3L y su interacción.
ESTRUCTURA SECUNDARIA
La estructura secundaria se puede observar en el siguiente enlace:
DOMINIOS
Existen 3 dominios de esta proteína:
● Dominio PWWP: se encarga de unir la proteína a la cromatina, en concreto a la heterocromatina pericéntrica; y por tanto resulta responsable de la metilación del DNA de esas zonas.
Está formado por 100-150 aminoácidos
y se caracteriza por una secuencia central con el motivo: Pro-Trp-Trp-Pro. Existen dos aminoácidos en la superficie del dominio, Lys y Arg, que son los sitios de unión a DNA.
Aparte de la unión a DNA, este dominio interacciona con otros factores, entre los que destacamos la Lys36 de la histona H3, fundamental para la unión a la cromatina; la proteína ZHX, cuya interacción con DNMT3-B contribuye
al silenciamiento de genes; y la timina-DNA-glicosilasa (TDG), que inhibe aparentemente la actividad DNA-metiltransferasa de DNMT3-A.
● Dominio ADD: con respecto a este dominio, su función principal es la unión a distintos factores.
La cola de la histona H3 se ajusta al surco menor del dedo PHD del dominio ADD, estableciéndose puentes de hidrógeno entre ambas moléculas, e induciendo un cambio conformacional de ADD. Lo que ocurre es que, inicialmente,
el dominio ADD está localizado en una región que no permite el acceso del DNA a la zona catalítica. Al unirse ADD a la histona, el cambio conformacional resultante permite el acceso del DNA.
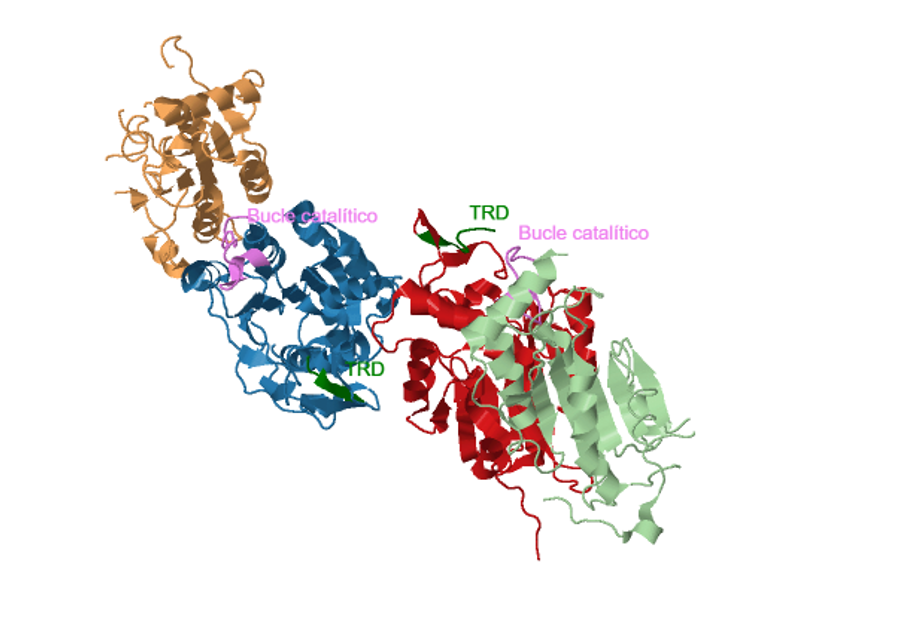
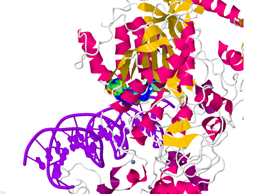
● Dominios C-terminales: “Dominio de reconocimiento de secuencia diana” (TRD), “bucle catalítico” e “interfaz homodimérica DNMT3A”. Todos ellos en su conjunto crean una superficie continua de unión a DNA.
CAMBIOS CONFORMACIONALES
El complejo DNMT3L - DNMT3A cambia su conformación según si está unido o no a DNA. Especialmente se da en los dominios TRD y en el bucle catalítico, del extremo C-terminal.
El dominio TRD se une al surco mayor del DNA, estableciendo contactos intermoleculares con el mismo. Adicionalmente, el TRD establece puentes de hidrógenos con hélices cercanas espacialmente, aumentando así la estabilidad
del complejo.
En cuanto al bucle catalítico, se une al surco menor del DNA.
Resulta preciso señalar que DNMT3L, aunque no se une al DNA, se une a DNMT3A en regiones próximas a zonas de unión a DNA, por lo que se confirma así sus papel regulador y estabilizante del proceso.
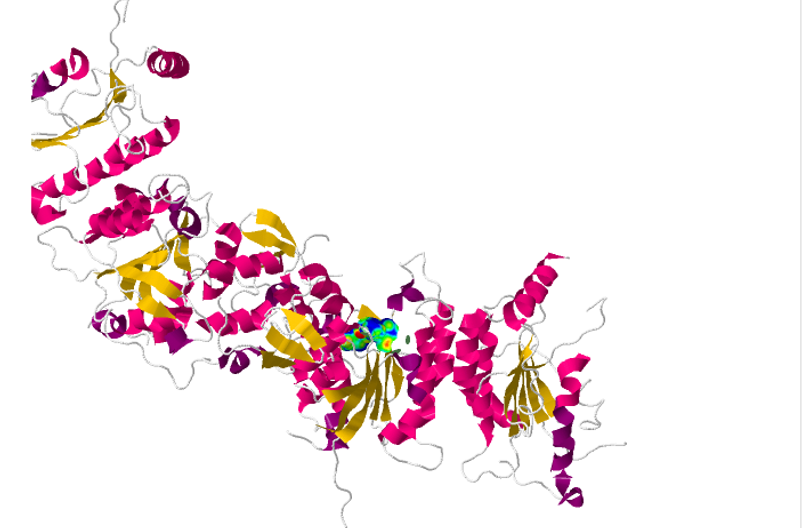
DNMT3 interacciona con sus ligandos por los residuos de Thr 641, Glu 660, Val 683, Trp 889. A continuación, se muestra la interacción de esta proteína con un análogo de sustrato, la S-Adenosil Homocisteína.
ASPECTOS INTERESANTES
Como aspecto interesante cabe indicar brevemente el mecanismo de reconocimiento de los nucleótidos susceptibles de ser metilados. Los procesos de metilación en epigenética se dan con una elevada frecuencia en los sitios CpG. Se
trata de regiones del DNA donde una citosina es seguida de una guanina, y son las citosinas de este dinucleótido las que pueden ser metiladas a 5-metilcitosina. El reconocimiento de este dinucleótido por parte de la enzima se da
gracias a múltiples interacciones entre el extremo C-terminal de la enzima con este dinucleótido (enlaces de hidrógeno y contactos de Van Der Waals) o con zonas adyacentes al mismo (interacciones electrostáticas y puentes de hidrógeno).
2. DNMT1:
ESTRUCTURA GLOBAL
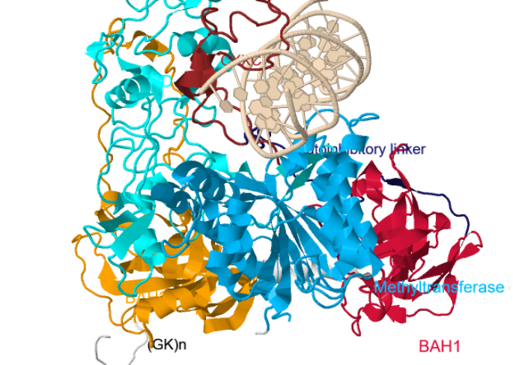
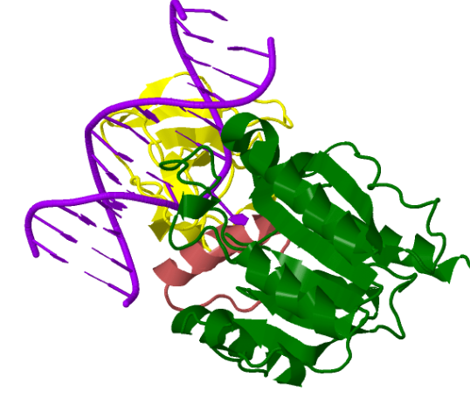
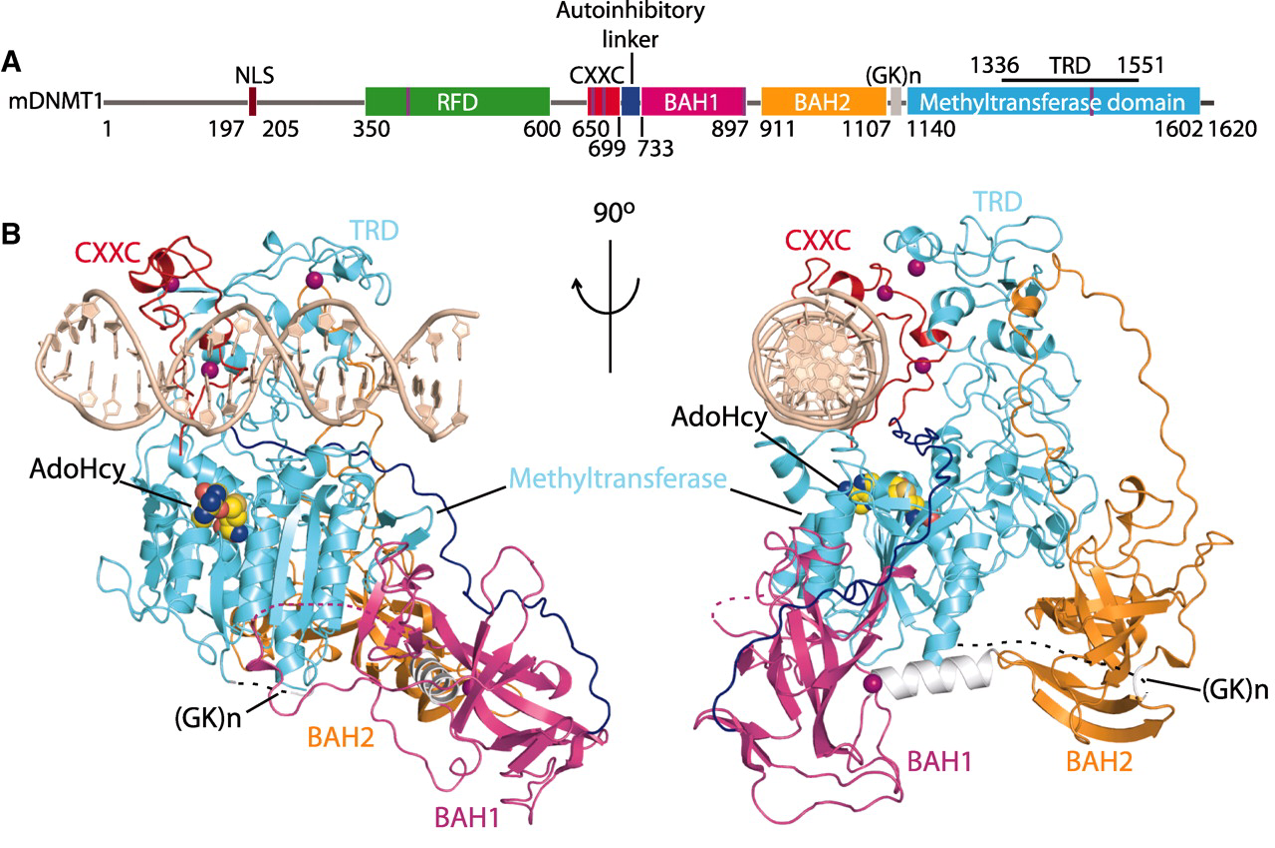
La metiltransferasa de mantenimiento de ADN (DNMT1) es una proteína multimodular compuesta por un dominio RFD (del inglés “replication fork-targeting domain”), un dominio CXXC de unión al ADN, un par de dominios BAH (del inglés “bromo-adjacent homology”) y un dominio catalítico C-terminal.
La DNMT1 presenta en su estructura secundaria todo tipo de estructuras secundarias, desde alfa hélices y hélices 310, hasta láminas beta y puentes beta.
Los dominios CXXC y BAH1 están en los extremos opuestos del dominio de metiltransferasa y están conectados por un segmento enlazador largo (enlazador CXXC-BAH1). Los dominios BAH1 y BAH2 están separados por un conector
α-helicoidal, con ambos dominios BAH posicionados en la superficie alejados del ADN unido. El segmento enlazador (GK) en que conecta el dominio BAH2 con el dominio catalítico está desordenado en el complejo y se muestra mediante
una línea discontinua. Observamos cuatro cationes Zn2+ en la estructura del complejo, dos en coordinación Cys4 dentro del dominio CXXC, mientras que otros dos implican la coordinación de iones Zn individuales en coordinación Cys3His
en BAH1 y en el dominio de reconocimiento (TRD) de la metiltransferasa.
DOMINIOS Y CAMBIOS CONFORMACIONALES
El dominio CXXC se une específicamente al dinucleótido CpG no metilado y coloca el conector CXXC-BAH1 entre el ADN y el sitio activo de DNMT1, evitando la metilación de novo. Además, un bucle que se proyecta desde BAH2 interactúa con el dominio TRD de la metiltransferasa, estabilizando la TRD en una posición retraída y evitando que se inserte en el surco principal del ADN.
El dominio de metiltransferasa de mDNMT1 se pliega en dos subdominios, denominados núcleo catalítico y TRD, que están separados por una gran hendidura que está ocupada por el ADN. El núcleo catalítico de DNMT1 está unido
por una hoja β antiparalela bicatenaria del dominio BAH1. El subdominio TRD se inserta entre la lámina β central y las últimas tres hélices α del núcleo catalítico. El dominio TRD se pliega en una unidad estructural independiente
y se estabiliza en parte por un ion Zn2+ coordinado por Cys3His. Además, un pliegue en forma de horquilla al comienzo del dominio TRD forma contactos hidrófobos con el núcleo catalítico y el dominio BAH1.
Los dominios BAH1 y BAH2 de mDNMT1 están conectados por una hélice α y están dispuestos en una configuración en forma de pesa. A pesar de la baja conservación de la secuencia ambos dominios BAH adoptan un tipo de plegamiento
común. Además, un Zn2+ coordinado por Cys3His ancla el dominio BAH1 a la hélice α enlazante. Ambos dominios BAH están físicamente asociados con el dominio metiltransferasa. En el dominio BAH2, un bucle largo se proyecta hacia
arriba y está anclado cerca de su punta al TRD del dominio de metiltransferasa. Esta interacción previene la interacción del TRD con el ADN. Los dominios BAH1 y BAH2 presentan una gran área de superficie accesible que es periférica
al dominio catalítico y podría servir como plataforma para las interacciones con otras proteínas. El dominio catalítico forma el núcleo del complejo y contacta tanto los dominios BAH como el ADN. Una molécula de AdoHcy se coloca
en el sitio activo del dominio catalítico.
3. DNA METILTRANSFERASA DE BACTERIAS (HhaI):
ESTRUCTURA GLOBAL
Es una citosina-5-metiltransferasa que forma parte del sistema de restricción-modificación de bacterias. Es una proteína muy sencilla con solo una subunidad.
ESTRUCTURA SECUNDARIA
La proteína presenta un porcentaje mayor de hélices alfa y 310 que de hebras beta.
DOMINIOS
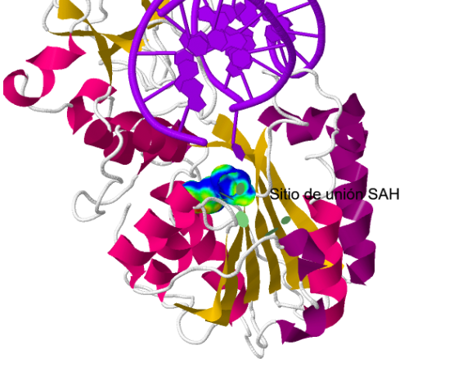
La estructura se pliega en dos dominios separados por una gran hendidura donde se encuentra el sitio de unión a ADN. El dominio grande proporciona el sitio de unión al cofactor (S-Adenosil-Met o S-Adenosil-HomoCys) y los residuos catalíticos. El dominio pequeño posee una región de residuos variable que se responsabiliza del reconocimiento específico del ADN. Presenta un bucle de sitio activo formado por 20 residuos que se encuentra en el dominio grande.
CAMBIOS CONFORMACIONALES
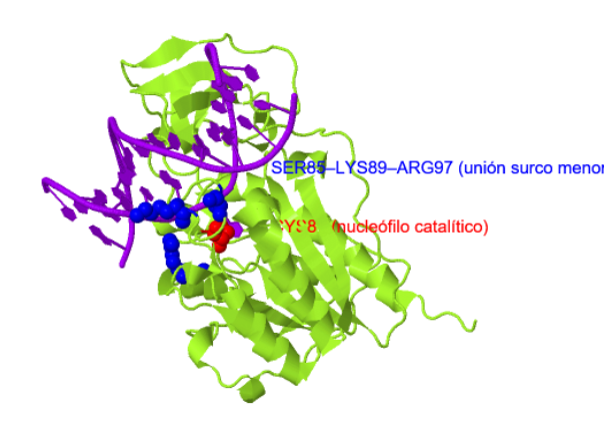
El bucle contiene seis residuos muy conservados incluyendo el nucleófilo catalítico Cys81. Al unirse al ADN, el bucle sufre un cambio conformacional y se mueve hacia la hendidura. Se pone en contacto con el surco menor del ADN con
3 residuos (Ser85, Lys89 y Arg97) que interaccionan con el esqueleto del monosacárido y fosfato. El grupo sulfhidrilo de Cys81 se acerca a la citosina y se forma un enlace covalente. El dominio pequeño de la proteína sufre
también un pequeño movimiento hacia la hendidura de unión al ADN.
El donador de grupos metilos principal de esta proteína es S-Adenosil-Homocisteína (SAH) que funciona como cofactor y se une al dominio grande.
Autores
Guion elaborado por Ana Sánchez Cruz, Eva Vicente López, Joel Pablos Martín, Jorge Úbeda Garrido y Carla Giménez Murcia, alumnos de Química e Ingeniería de Proteínas, 3er curso del Grado en Biotecnología, curso 2019/20.
Referencias
1. Wu J. C., Santi D. V. (1987) Kinetic and Catalytic Mechanism of HhaI Methyltransferase. The Journal of Biological Chemistry. Vol: 262, No. 10, pp. 4778-4786
2. Klimasauskas S., Kumar S., Roberts R.J., Cheng X. (1994) Hhal Methyltransferase Flips Its Target Base Out of the DNA Helix. Cell, ISSN: 0092-8674, Vol: 76, Issue: 2, Page: 357-69
3. Cheng X., Kumar S., Posfai J., Pflugrath J. W., Roberts R.J. (1993) Crystal Structure of the Hhal DNA Methyltransferase Complexed with S-Adenosyl-L-Methionine. Cell, ISSN: 0092-8674, Vol: 74, Issue: 2, Page: 299-307
3. Cheng X., Kumar S., Posfai J., Pflugrath J. W., Roberts R.J. (1993) Crystal Structure of the Hhal DNA Methyltransferase Complexed with S-Adenosyl-L-Methionine. Cell, ISSN: 0092-8674, Vol: 74, Issue: 2, Page: 299-307
4. Zhang et al. (2018) Structural basis for DNMT3A-mediated de novo DNA methylation. Nature 15, 554 (7692): 387-391.
5. Rodríguez Dorantes, M., Téllez Ascencio, N., Cerbón, M. A., López, M., & Cervantes, A. (2004). Metilación del ADN: un fenómeno epigenético de importancia médica. Revista de investigación clínica, 56(1), 56-71.