Introducción
La formación de puentes disulfuro es un proceso clave en el correcto plegamiento de las proteínas. En el retículo endoplásmico (RE) de eucariotas, la formación de puentes disulfuro está catalizada por las enzimas Proteína Disulfuro Isomerasas,
PDI, siendo análogas a las proteínas Dsb (del inglés Disulfide bridge-forming enzymes), que son oxidorreductasas que actúan en el periplasma de procariotas.
En eucariotas, la mayoría de las proteínas se pliegan en la luz del RE (unos de los pocos lugares en los que se pueden formar puentes disulfuro), donde hay chaperonas, peptidilprolil cis-trans isomerasas, PDI y otras enzimas que
llevan a cabo modificaciones postraduccionales. La PDI, además de formar puentes disulfuro de novo, puede corregir puentes disulfuro erróneos. A mayores, puede actuar como una chaperona. La familia PDI es la familia de proteínas
más abundante en células de mamíferos y levaduras.
Estructura
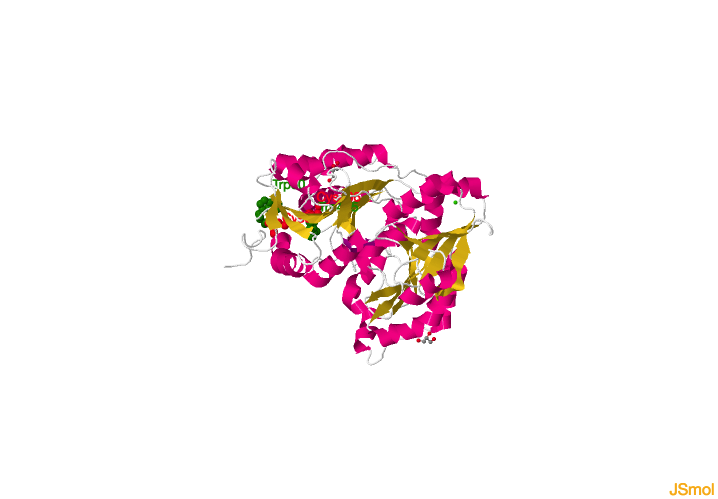
Las PDIs pertenecen a la superfamilia tiorredoxina (TRX), caracterizada por la presencia de dominios tipo tiorredoxina (TXR-like) con similitud estructural con la TRX.
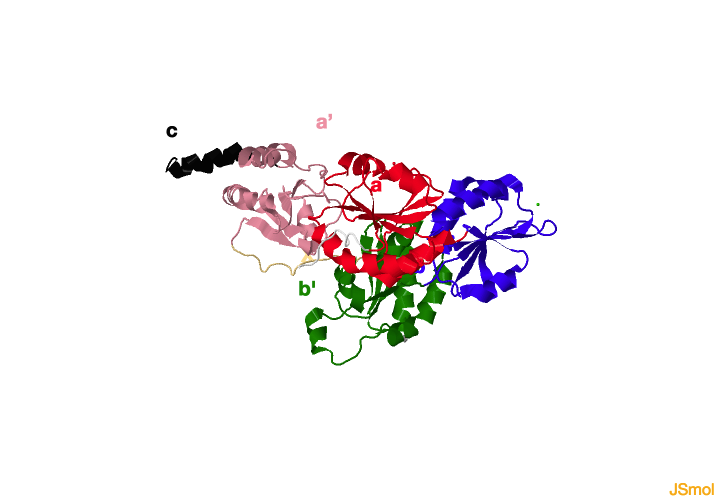
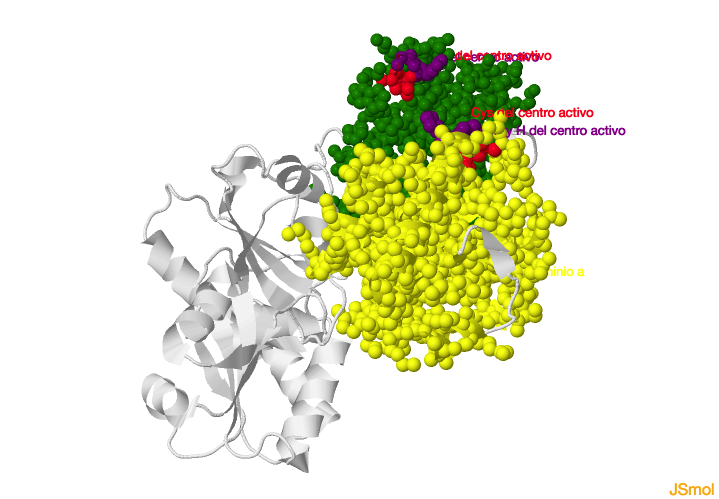
La estructura de la PDI se organiza en cuatro dominios denominados
a,
b,
b’ y
a’ que adquieren una conformación en forma de “U”. Situado entre los dominios b’ y
a’ hay un lazo o
conector (X) y un dominio C-terminal (c) a continuación del dominio a´. La organización global de la proteína es abb'Xa’c
Estructura de la PDI de levadura.
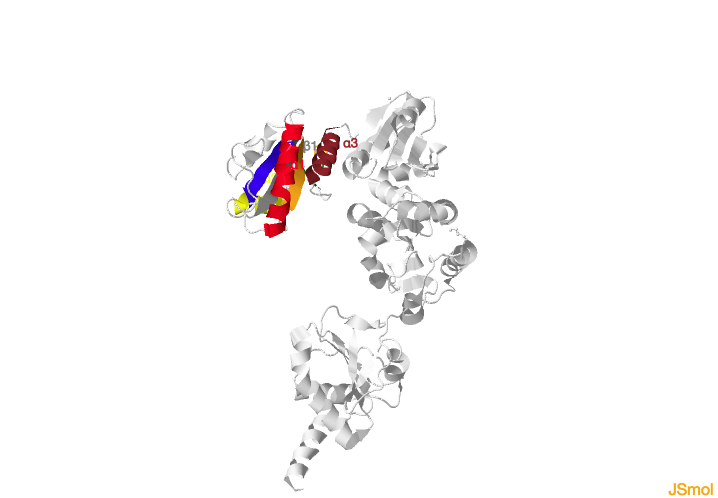
Los dominios a, a´, b y b´ son dominios tipo tiorredoxina que se pliegan con el siguiente motivo:
β1
α1
β2
-
α2
-
β3
β4
α3
como puede observarse en el siguiente enlace.
Motivos de plegamiento del dominio tipo TRX β1α1β2-α2-β3β4α3.
De los cuatro dominios tipo TRX, solo a y a´ poseen un motivo catalítico
CXXC en su sitio activo, en el que los dos aminoácidos intermedios son claves para determinar el potencial redox, siendo XX: GH.
El
motivo CXXC se encuentra en el N-terminal de la primera hélice
α1. En la PDI de levadura mostrada en el enlace inferior, se resaltan en azul los residuos
Cys61-Gly62-His63-Cys64 y
Cys406-Gly407-His408-Cys409 de los motivos CXXC de a y a´ respectivamente. También todos los dominios tipoTRX tienen un bucle
cis-Pro que precede al motivo
β3
β4
α3 y permite interaccionar con otras proteínas. Además, permite reducir el pKa de la
cisteína N-terminal del motivo CXXC de tal manera que a pH fisiológico se desprotona. Por otra parte, el
Glu30 en el dominio a y el
Glu372 del dominio a’ presentes en la hebra
β1 intervienen también en el proceso redox.
El dominio c está compuesto sobre todo por aminoácidos cargados negativamente. Este dominio tiene una hélice α que parece estabilizar el dominio a’, aunque no parece intervenir en el mecanismo catalítico.
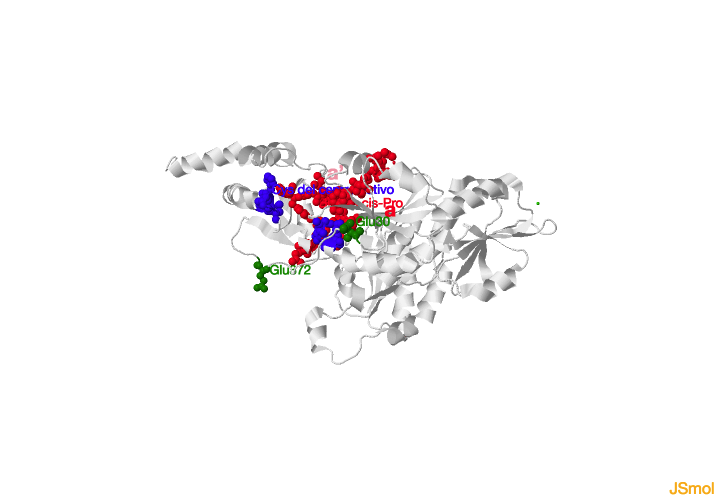
Residuos claves en el mecanismo de la PDI.
Mecanismo de acción
Recordemos que la PDI lleva a cabo dos tipos de reacciones: corrección de puentes disulfuro erróneos, y formación de puentes disulfuro correctos. En el proceso de revertir puentes disulfuro erróneos, es necesario que la PDI
esté en su estado reducido, aunque no se sabe si tienen que estar los dos dominios redox de a y a´ reducidos.
Estructura de la PDI en su estado reducido.
La reacción es iniciada por un ataque nucleofílico del tiolato expuesto a un puente disulfuro no nativo, formando un intermediario con un puente disulfuro entre la PDI y la proteína mal plegada. El otro tiolato de la proteína mal
plegada ahora puede generar un puente disulfuro con otra cisteína, en este caso con una cisteína correcta. El puente disulfuro entre las dos proteínas se rompe gracias al otro tiolato de la PDI (Figura 1).
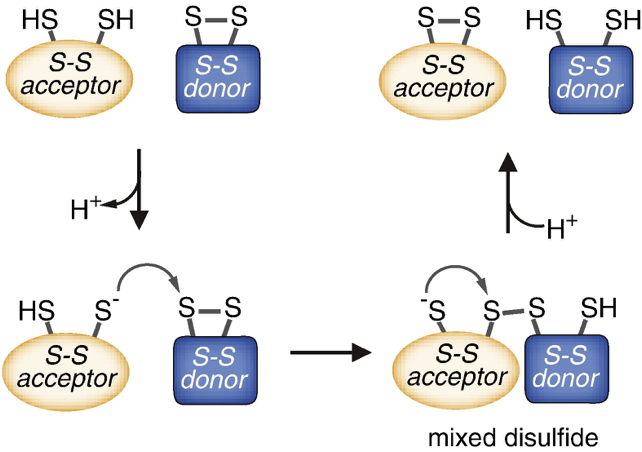 Figura 1: Mecanismo de intercambio de puentes disulfuro. Tomado de Appenzeller-Herzog & Ellgaard, 2008.
Figura 1: Mecanismo de intercambio de puentes disulfuro. Tomado de Appenzeller-Herzog & Ellgaard, 2008.
En la síntesis de novo de puentes disulfuro, la PDI activa está oxidada.
Estructura de la PDI en su estado oxidado.
Después de la catálisis, la PDI se ha reducido para oxidar dos cisteínas de la proteína sustrato por lo que es necesaria la intervención de la flavoproteína Ero1 (ER oxidoreductin 1), que transfiere equivalentes oxidantes a
la PDI para recuperar su estado redox activo (Figura 2 y Figura 3).
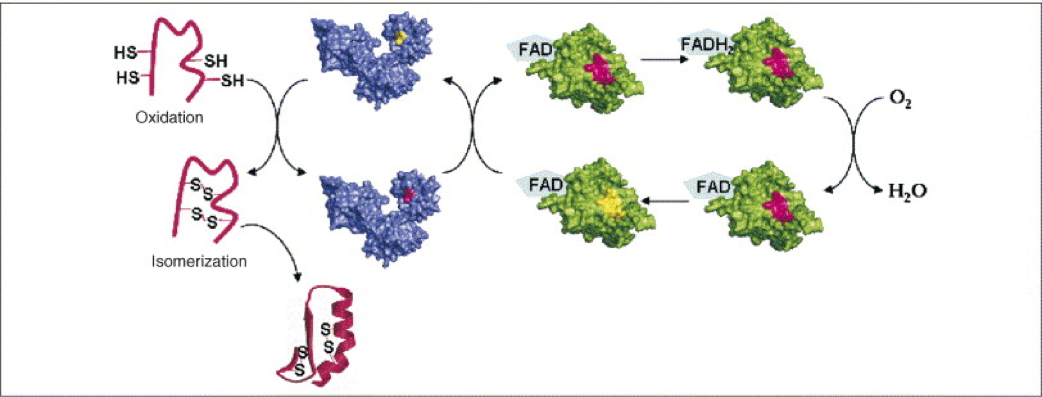 Figura 2: Mecanismo de síntesis de novo de puentes disulfuro. Tomado de Gruber et al., 2006.
Figura 2: Mecanismo de síntesis de novo de puentes disulfuro. Tomado de Gruber et al., 2006.
Ero1 transfiere los electrones desde la PDI reducida hasta el aceptor final de electrones, que suele ser el oxígeno molecular, que se reduce a H2O2.
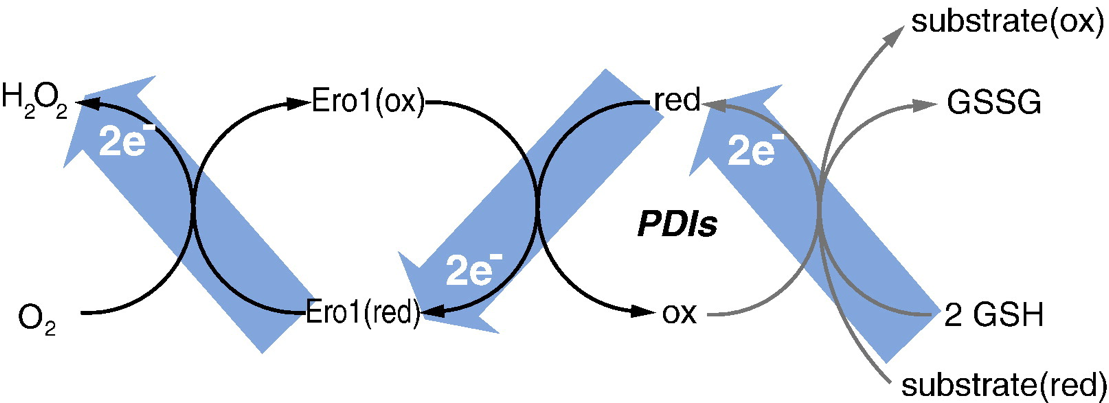 Figura 3: Flujo de electrones entre la PDI, la proteína sustrato y Ero1. GSH y GSSG son glutatión reducido y oxidado respectivamente. Tomado de Appenzeller-Herzog & Ellgaard, 2008.
Figura 3: Flujo de electrones entre la PDI, la proteína sustrato y Ero1. GSH y GSSG son glutatión reducido y oxidado respectivamente. Tomado de Appenzeller-Herzog & Ellgaard, 2008.
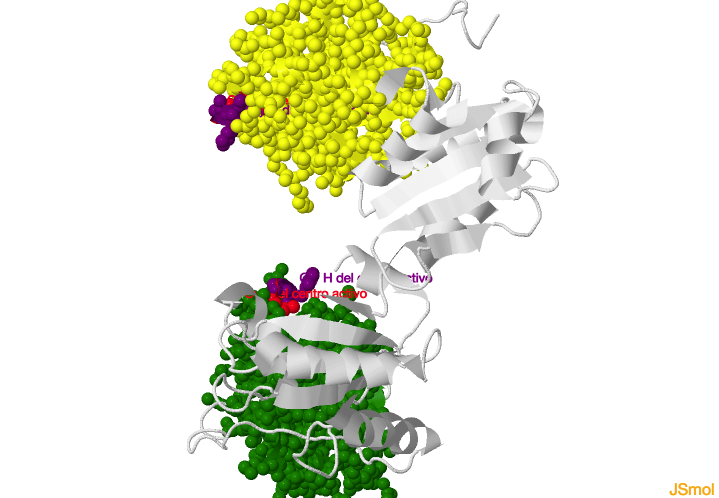
La unión del sustrato se cree que se realiza en la cavidad U. Algunos estudios indican que la región C-terminal del dominio a’ y los primeros residuos del dominio c son los más importantes para la unión del sustrato. No obstante,
b’ también puede unir pequeños péptidos. Aunque la PDI es una proteína soluble, presenta parches hidrofóbicos en a y a’, además de en b y b’, que serían importantes para unir proteínas desplegadas o parcialmente desplegadas,
por lo que presentan residuos hidrofóbicos expuestos en la superficie. Como ya se ha dicho antes, la PDI también puede actuar como chaperona, siendo importantes en este caso los dominios a’ y c.
Los dos sitios activos se
enfrentan en la U, y se crea un bolsillo hidrofóbico lo suficientemente grande como para acomodar 100 residuos de la proteína sustrato. La mayor diferencia entre los sitios activos a y a’ es el estado de oxidación de
sus cisteínas. Los dominios a’ están en su estado reducido y los dominios a pueden estar oxidados (80%) o reducidos (20%).
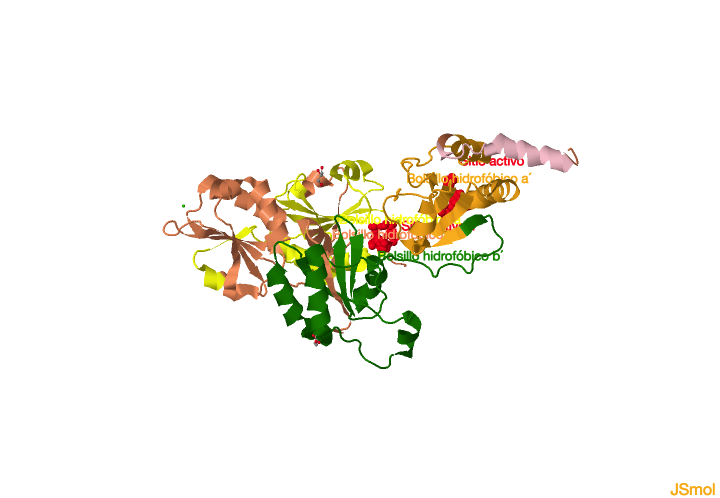
Los dos sitios activos se enfrentan en la conformación en U.
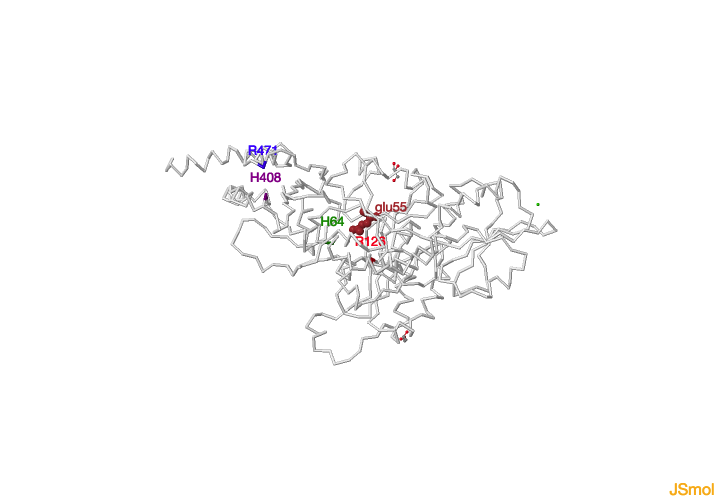
La mayoría de los residuos cercanos a las cisteínas de los sitios activos son idénticos en ambos dominios, como los residuos H64 y R126 de a y H408 y R471 de a’, que regulan el potencial redox. No obstante, por ejemplo,
E55 y Q87 del dominio a se cambian por L400 y K434 de a’. E55 se cree que es útil para facilitar la salida del sitio activo. En el dominio a’ no existe dicho mecanismo de salida del sitio activo, ya que se sustituye
por L400, aunque es cierto que L400 explica por qué a es más activo en su forma oxidada.
Los residuos adyacentes al sitio activo H64 , R126 ,H408 y R471 son importantes para la regulación del potencial redox.
En cuanto al acceso al sitio activo, en el dominio a hay un triptófano,
W60, próximo al sitio activo que presenta poca movilidad, ya que interacciona con el dominio b; sin embargo, en la subunidad a’ el
W405 sí que es muy móvil y no participa en ninguna interacción. Además,
C406 está más expuesta al solvente que
C61 debido a estos cambios del triptófano; no obstante, en ambos casos este triptófano es importante para interaccionar con el substrato.
Residuos importantes para el acceso al sitio activo W60 y W405.
El acceso al sitio activo también está facilitado por la glicina del motivo
C-G-H-C, ya que permite tomar ángulos de torsión alcanzables por otros residuos, además de que al carecer de cadena lateral permite la acomodación de diferentes sustratos.
Guion elaborado por Guillermo Martín Ramos, Francisco Javier Palomero Hernández, Enrique Presa Díez y Diego Sainz García, alumnos de Química e Ingeniería de Proteínas, 3er curso del Grado en Biotecnología, curso 2017/18.
Cuestiones
Responder al cuestionario correspondiente en la pagina de Studium
Referencias
Appenzeller-Herzog, C. & Ellgaard, L. (2008). The human PDI family: Versatility packed into a single fold. BBA 1783, 535–548.
Gruber, C.W.; Čemažar, M.; Heras, B.; Martin, J.L. & Craik D.J. (2006). Protein disulfide isomerase: the structure of oxidative folding. Trends Biochem Sci. 31(8), 455-64.
PDB www.rcsb.org, H.M. Berman, J. Westbrook, Z. Feng, G. Gilliland, T.N. Bhat, H. Weissig, I.N. Shindyalov, P.E. Bourne (2000) The Protein Data Bank URL: Nucleic Acids Research, 28, 235-242.
Tian, G.; Xiang, S.; Noiva, R.; Lennarz, W.J. & Schindelin, H. (2006). The crystal structure of yeast protein disulfide isomerase suggests cooperativity between its active sites. Cell 124(1), 61-73.