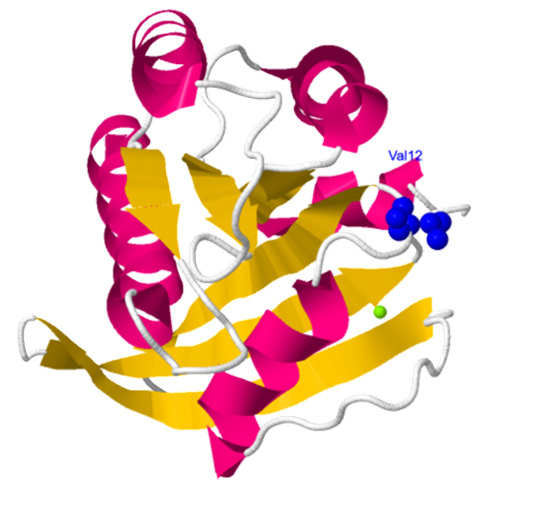Definición y función:
Las proteínas RAS fueron identificadas en un estudio de oncogenes retrovirales productores de sarcomas en ratas y desde los años 80, se ha llevado a cabo un intenso estudio de su estructura y bioquímica debido a que se han encontrado
mutaciones en RAS al analizar células tumorales humanas. Estas mutaciones en RAS dan lugar a formas de RAS activas constitutivamente.
Las proteínas RAS son GTPasas de bajo tamaño molecular localizadas en la cara interna de la membrana plasmática y que pueden pasar del estado activado (unidas a GTP) al estado inactivado (unidas a GDP). El paso entre ambos está mediado por GEFs (guanine
nucleotide exchange factors) y GAPs (GTPasa-activating proteins). Este mecanismo de hidrólisis de GTP a GDP está altamente conservado, pero cuando se producen mutaciones activadoras de RAS, la proteína se mantiene en su forma constitutivamente
activa. Esto puede dar lugar a RASopatías, cáncer y algunos desórdenes psiquiátricos como el autismo.
Las rutas de señales que activan a RAS son muy diversas e incluyen receptores tirosina quinasas, receptores acoplados a proteínas G, integrinas y receptores de citocinas.
Existen tres genes que codifican RAS (H-, N- y K-RAS) y se ha podido observar su implicación en el desarrollo de diferentes tipos de tumores cuando se dan mutaciones activadoras de RAS. Las mutaciones en el gen K-RAS son las más tumorigénicas, con incidencias
muy altas en cáncer de pulmón, páncreas y colon. Las mutaciones en N-RAS producen un 30% de las leucemias agudas no linfoides y H-RAS es el de menor incidencia en cáncer, aunque se han encontrado mutaciones en H-RAS en cáncer de vejiga,
próstata y tiroides.
Estructura general de RAS:
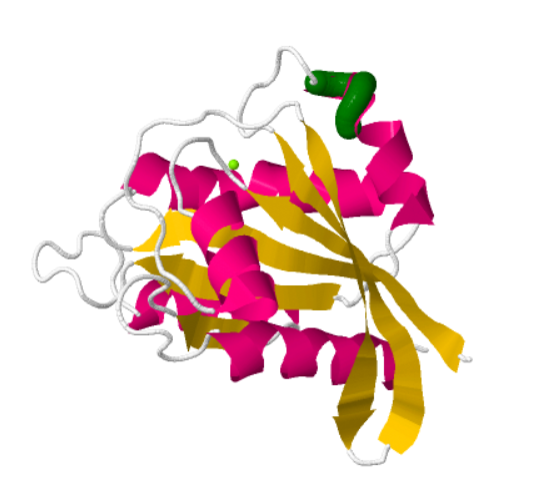
La proteína H-RAS posee en total 166 residuos; formando siete hélices α, que suman 70 residuos en total, y seis hebras β, que suman 44 residuos. Además, posee nueve lazos entre estos dos tipos de estructura secundaria. Cuatro lazos,
entre los que destacan el lazo P o las regiones Switch I y Switch II, se encargan de que la proteína pueda unirse a GDP. En la estructura de RAS destaca también una vuelta de hélice 310.
Estructura genaral de la proteína RAS: En la estructura PDB se pueden observar las hélices alfa en rosa, las hebras beta en amarillo y la vuelta de hélice 310 en verde. Los lazos se observan en color blanco.
La proteína H-RAS funcionalmente posee dos dominios. Además del dominio catalítico (compuesto por 166 residuos y con una estructura anteriormente mencionada), existe un dominio de unión (formado por 12 residuos). Para el anclaje a la membrana, las proteínas
RAS presentan un ancla lipídica (compuesta por farnesilo o hexadecilo, entre otras moléculas lipídicas), unida covalentemente a residuos del dominio de unión. El dominio de unión y la zona del ancla lipídica se denominan en conjunto
como Región C-terminal hipervariable, que posee estructura de “coil-bend-coil”. La estructura se estabiliza gracias a la presencia de un puente salino entre un residuo del dominio de unión y el residuo 49 de la hebra β-2 de RAS.
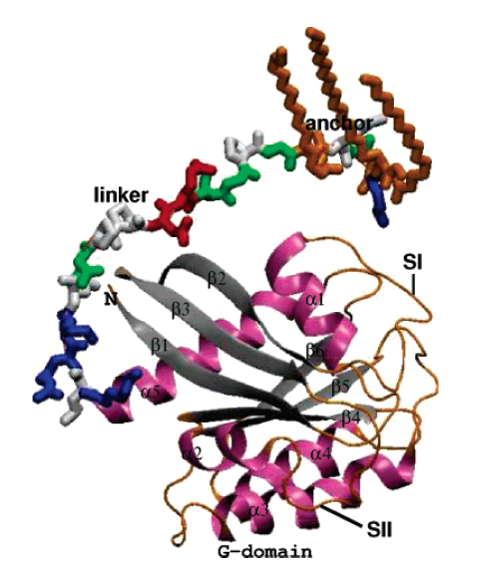 Estructura general de la proteína RAS con el ancla lipídica: En la imagen se puede observar el dominio de unión (“linker”) y el ancla lipídica (“anchor”).
Estructura general de la proteína RAS con el ancla lipídica: En la imagen se puede observar el dominio de unión (“linker”) y el ancla lipídica (“anchor”).
Las tres isoformas de RAS poseen una conformación similar. Los primeros 86 aminoácidos son iguales en las tres proteínas; mientras que la identidad de los residuos 87-166 es de un 85%. La Región C-Terminal hipervariable está presente en las tres proteínas,
pero varía en cada una de ellas.
Estructura de dominios importantes:
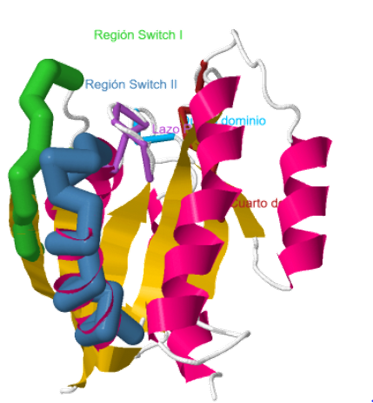
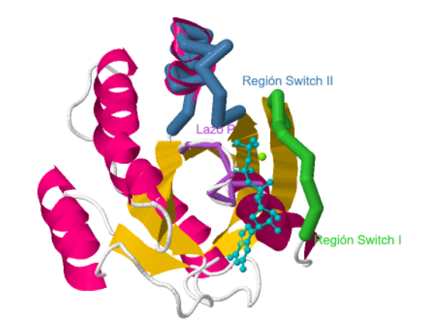
Podemos diferenciar 5 dominios de importancia en esta familia de proteínas.
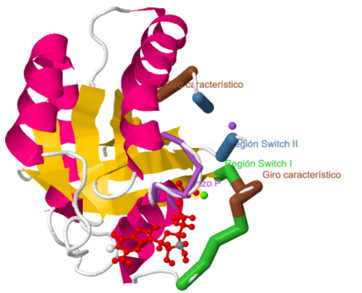
El primero de los dominios conocidos es el p-loop. Este es un dominio de gran importancia ya que regula la interacción y unión con el fosfato β del GDP o GTP. Está formado por los aminoácidos 10-17.
El segundo dominio, también conocido como región Switch I, está formado por los aminoácidos 30 – 40. Los residuos que interaccionan con el grupo fosfato son Tyr32 y Thr35. Este último es el de más importancia, ya que une el fosfato terminal del GTP y
el ion divalente de magnesio unido al sitio activo.
El tercer dominio, conocido como región Switch II, está compuesto por los aminoácidos 59 – 76. Este dominio está localizado entre la lámina β central de la proteína RAS y la hélice α2. El residuo que interacciona con el grupo fosfato es Gly60. La región
switch II es un dominio DXXGQ. El residuo D corresponde a aspartato 57, específico para la unión con guanina en lugar de adenina (por ello la unión con GTP y no ATP). El residuo Q corresponde a glutamina 61, el residuo crucial que
activa la molécula de agua que va a realizar la reacción catalítica necesaria para la hidrólisis de GTP a GDP.
Dominios característicos de la proteína RAS: En la estructura PDB se pueden observar el dominio P-loop o lazo P (en morado), la región Switch I (en verde) y la región Switch II (en azul oscuro). El cuarto dominio se muestra en color marrón y el quinto en color azul claro.
El cuarto dominio presenta una secuencia primaria LVGNKxDL, que también favorece la interacción específica de la proteína con guanina.
El quinto dominio contiene una secuencia consenso SAK. El residuo A corresponde a alanina 146, que permite un aumento de la especificidad por guanina en lugar de adenina.
El segundo y tercer dominio, o regiones Switch, constituyen la parte principal de la proteína. Sufren cambios conformacionales cuando se produce la activación de las RAS por unión a GTP. Este cambio conformacional en los dos dominios es el que principalmente
atribuye a las proteínas RAS su función básica como proteínas de intercambio.
Regulación y rutas de transducción
La proteína RAS tiene un papel muy importante en las rutas de transducción de señales implicadas en el crecimiento y la diferenciación celular. Entre estas rutas de transducción en las que está implicada RAS se encuentra la ruta de las MAP quinasas, implicada
en la normal señalización de células eucariotas, pero que puede verse alterada fácilmente en células tumorales. Esta alteración de la ruta de transducción debida a cambios en la proteína RAS se traduce en un aumento de la actividad
de la proteína y, por tanto, una actividad mayor de la ruta.
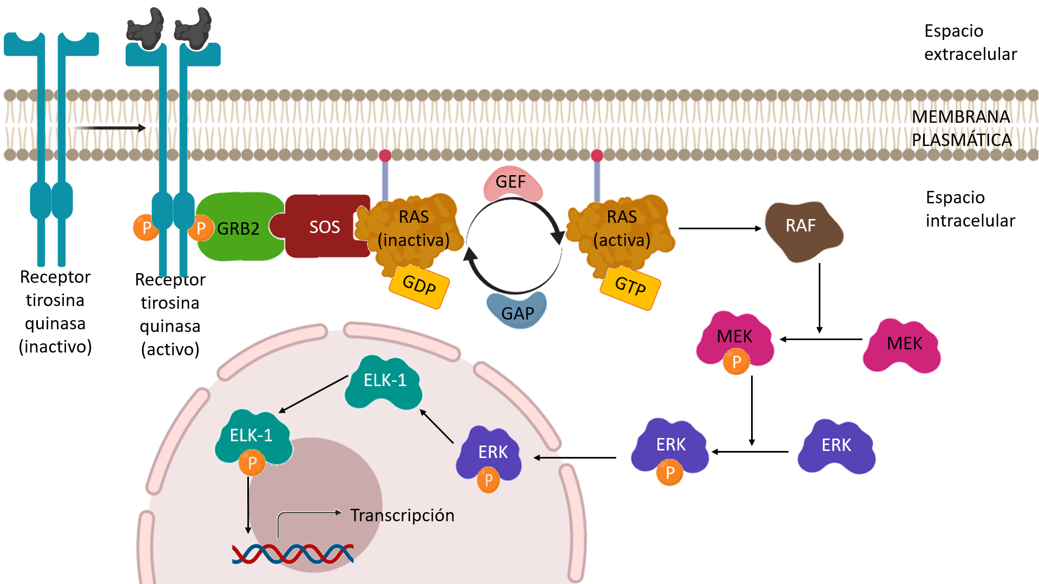 Ejemplo de ruta de transducción en las que participa RAS: La proteína RAS interviene en rutas de transducción mediadas por receptores como los receptores tirosina quinasa.
Ejemplo de ruta de transducción en las que participa RAS: La proteína RAS interviene en rutas de transducción mediadas por receptores como los receptores tirosina quinasa.
La conversión entre la forma de unión a GDP, estable e inactiva, a la forma de unión a GTP, activa y más inestable, es estimulada por factores de intercambio de nucleótidos de guanina (GEFs). Por otro lado, la vuelta a la forma de unión a GDP está regulada
por proteínas activadoras de la actividad GTPasa (GTPase activating proteins, GAPs). Estas proteínas, GEFs y GAPs, interaccionan con diferentes biomoléculas como lípidos u otras proteínas, regulando la función de la proteína RAS.
Unión de RAS a GTP y GDP: En la estructura PDB se puede observar la interacción entre la proteína RAS y una molécula de GTP (en color azul claro) o una molécula de GDP (en color rojo).
El hecho de que la activación o inactivación de RAS esté controlada por proteínas como las GEFs o GAPs permite que la regulación sea muy precisa y que RAS sea capaz de responder frente a diferentes estímulos. La regulación llevada a cabo por estas proteínas
se basa principalmente en la translocación, ya que activan o inactivan a RAS simplemente al situarse en sus proximidades.
La unión de GEF a RAS origina cambios conformacionales en las regiones Switch (Switch I y Switch II), al pasar RAS del estado activo al estado inactivo. También se producen cambios conformacionales en el lazo P, lo que disminuye la afinidad por GDP y
permite la unión de GTP.
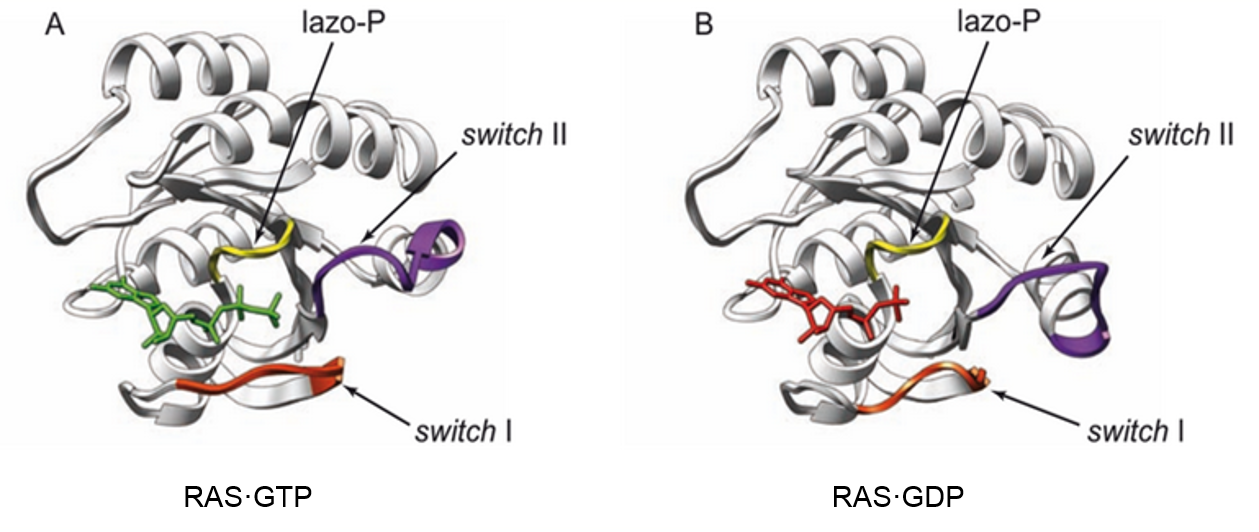 Cambios conformacionales de RAS: La interacción con GTP y GDP origina cambios conformacionales en RAS. La imagen A corresponde a RAS unida a GTP (estado activo) y la imagen B corresponde a RAS unida a GDP (estado inactivo).
Cambios conformacionales de RAS: La interacción con GTP y GDP origina cambios conformacionales en RAS. La imagen A corresponde a RAS unida a GTP (estado activo) y la imagen B corresponde a RAS unida a GDP (estado inactivo).
Mutaciones en esta proteína y cáncer:
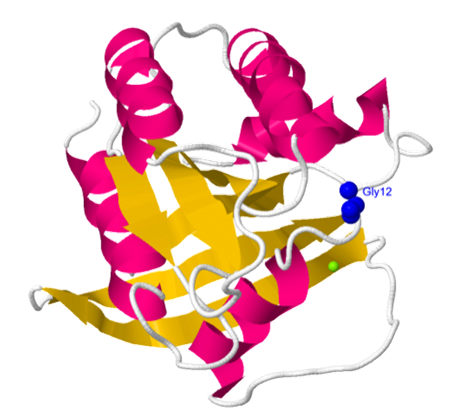
Los protooncogenes RAS pueden convertirse en oncogenes debido a mutaciones puntuales cuyas consecuencias son la sustitución de un aminoácido en posición clave, siendo las mutaciones de la G12 y la Q61 las más comunes.
Aproximadamente, el 30% de los cánceres están relacionados con mutaciones en la proteína RAS, que originan la disminución de la capacidad de hidrólisis de GTP y, por lo tanto, hacen que permanezca en su forma activa de forma constitutiva.
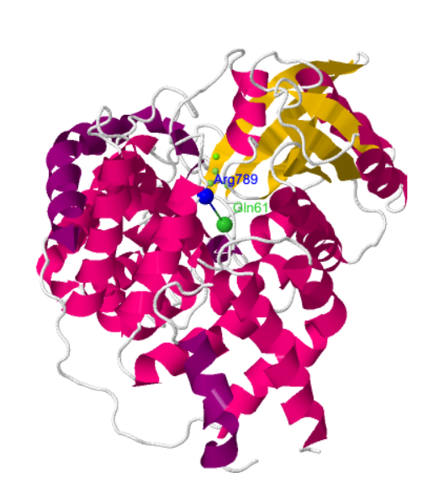
La mutación correspondiente al intercambio de glicina 12 por valina (mutación G12V) es muy importante porque el cambio sufrido por el residuo de glicina dificulta la interacción del residuo arginina 789 de la proteína GAP y el NH2 del residuo de glutamina
61 de RAS, impidiendo la hidrólisis de GTP por GDP por falta de puentes de hidrógeno estabilizadores de la reacción.
Mutación G12V: En la estructura PDB se puede observar el cambio de residuo de glicina a valina en la posición 12.
La mutación del residuo de glutamina 61, menos frecuente, origina que una molécula de agua no pueda posicionarse correctamente para permitir el ataque nucleofílico al γ-fosfato. Por tanto, la hidrólisis del GTP no es posible.
Interacción Q61-R789: En la estructura PDB se puede observar la interacción de RAS con GAP, concretamente de los residuos glutamina 61 (marcado en verde) y arginina 789 (marcado en azul). En la estructura se muestra el puente de hidrógeno que se establecería para favorecer
la hidrólisis de GTP.
La forma de RAS activa constitutivamente da lugar a la proliferación incontrolada de las células cancerígenas y la estimulación de la vía de quinasas MAP, también en ausencia de algunas hormonas o factores de crecimiento.
Autores
Guion elaborado por Ana Dávila Hidalgo, Daniel López de Prada, Álvaro García Rodríguez, Vincent Suerdieck y Lydia Iglesias Sánchez, alumnos de Química e Ingeniería de Proteínas, 3er curso del Grado en Biotecnología, curso 2019/20.
BIBLIOGRAFÍA
Ras Protein - an overview | ScienceDirect Topics. https://www.sciencedirect.com/topics/neuroscience/ras-protein.
Las rutas de señales mediadas por Ras. :: Centro de investigación del Cáncer - Comprehensive Cancer Center Research. http://www.cicancer.org/es/las-rutas-de-senales-mediadas-por-ras
Gorfe, A. A., Hanzal-Bayer, M., Abankwa, D., Hancock, J. F. & McCammon, J. A. Structure and Dynamics of the Full-Length Lipid-Modified H-Ras Protein in a 1,2-Dimyristoylglycero-3-phosphocholine Bilayer. J. Med. Chem. 50,
674–684 (2007).
Goodsell, D. S. Ras Protein. RCSB Protein Data Bank (2012). doi:10.2210/rcsb_pdb/mom_2012_4
Zhou, B., Der, C. J. & Cox, A. D. The role of wild type RAS isoforms in cancer. Semin. Cell Dev. Biol. 58, 60–69 (2016).
Han, C. W., Jeong, M. S. & Jang, S. B. Structure, signaling and the drug discovery of the Ras oncogene protein. BMB Rep. 50, 355–360 (2017).
Arias-Palomo, E. & Ernesto. Análisis Estructural de proteínas reguladoras de GTPasas de la superfamilia Ras mediante microscopía electrónica. (2008).
Pylayeva-Gupta, Y., Grabocka, E. & Bar-Sagi, D. RAS oncogenes: weaving a tumorigenic web. Nat. Rev. Cancer 11, 761–774 (2011).
Han, C. W., Jeong, M. S. & Jang, S. B. Structure, signaling and the drug discovery of the Ras oncogene protein. BMB Rep. 50, 355–360 (2017).
Simanshu, D. K., Nissley, D. V & McCormick, F. RAS Proteins and Their Regulators in Human Disease. Cell 170, 17–33 (2017).
Scheffzek, K. The Ras-RasGAP Complex: Structural Basis for GTPase Activation and Its Loss in Oncogenic Ras Mutants. Science (80-. ). 277, 333–338 (1997).