Definición y función:
La proteína humana p53 es un factor de transcripción que actúa de manera crucial en el control de la proliferación celular y en la inducción de la respuesta celular ante daño en el DNA. La importancia de esta proteína radica
en que actúa como un agente supresor de tumores, ya que detiene el ciclo celular en caso de mutación; si el daño es leve, detiene el ciclo celular para iniciar la reparación, mientras que si es grave, induce apoptosis en la
célula. Por esta razón, p53 se conoce a su vez como el “guardián del genoma”.
El gen p53, localizado en el brazo corto del cromosoma 17, codifica la proteína homónima que se encuentra en todos los tejidos humanos. Su nombre deriva de su peso molecular, que aunque realmente es 43.7kDa,
la proteína tiene un peso molecular aparente de 53kDa, y en un gel de electroforesis migra como tal.
Como factor de transcripción, tiene dominios de unión al DNA que hacen que regule la transcripción de ciertos genes dependiendo de las señales citoplasmáticas presentes en la célula. Además se ha visto que es el gen que más
frecuentemente muta en los tumores humanos, corroborando su papel central en el control de este tipo de patologías.
Por otro lado, estudios recientes han demostrado que mutaciones en el gen p53 pueden dar lugar a una ganancia de función que acelera la proliferación y expansión de los tumores, en contra de lo que tradicionalmente
se había tenido en consideración acerca de esta proteína.
Estructura general de p53
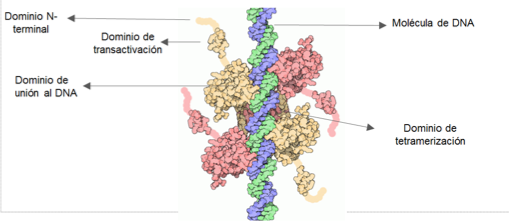
p53 es una proteína homotetramérica, donde cada monómero de 393 aminoácidos se divide en distintos dominios funcionales: dominio amino terminal, dominio de unión a DNA, dominio de tetramerización, y dominio carboxilo terminal, como se muestra en la figura
1.
 Union de p53 al DNA
Union de p53 al DNA
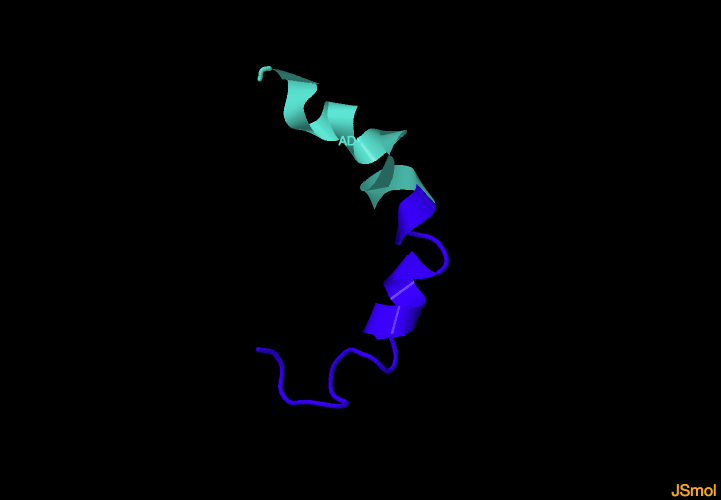
Dominio amino terminal (N-terminal)
Los primeros 94 aminoácidos de p53 contienen dos dominios de transactivación (AD1 y AD2), una región rica en prolinas, una señal de exportación nuclear (NES) y múltiples sitios que, ante una señal de estrés, sufren modificaciones
postraduccionales (fosforilaciones). Los dominios de transactivación
AD1 (20-40, en turquesa) y AD2 (40-60) son imprescindibles para la activación transcripcional de genes diana, mientras que la región rica en prolinas (60-90), que contiene cinco copias de la secuencia PXXP,
está implicada en apoptosis regulada por p53.
Se trata de un dominio de vital importancia para la activación de la proteína, ya que cuando se fosforila el N-terminal ésta se libera de MDM2, su regulador celular natural.
Fragmento de extremo N- terminal, conteniendo parte de AD1 (residuos 20-32)
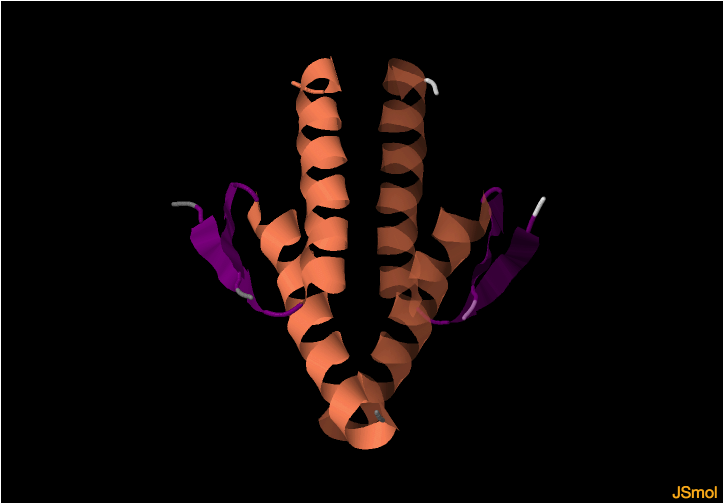
Dominio de tetramerización (Tet)
Este dominio está localizado entre los aminoácidos 320 - 360 y es responsable de la formación del tetrámero. Mediante cristalografía y RMN se ha visto que está formado por
dos hebras β (en morado) y dos
α-hélices entrecruzadas (en naranja).
Una región flexible de unos 30 aminoácidos es la encargada de unir este dominio con el dominio de unión a DNA.
Dominio de tetramerización
Dominio carboxilo terminal (C-terminal) o dominio básico
Este dominio comprende los aminoácidos 363-393. Contiene varios sitios que son modificados postraduccionalmente ante señales de estrés, como ubiquitinación, sumolización, fosforilación, acetilación o glicosilación.
El C-terminal, gracias a su abundancia en residuos básicos de arginina y
lisina (en morado), confiere un punto isoeléctrico de 10. De esta manera es capaz de unir DNA de forma inespecífica mediante interacciones electrostáticas. Esta capacidad le permite regular la actividad del dominio de unión
a DNA.
Se ha resuelto mediante RMN una región del dominio (367-386) apenas estructurada.
No se ha conseguido cristalizar este dominio.
Fragmento de C-terminal, residuos 382-393
Estructura del dominio de unión a DNA y aminoácidos implicados
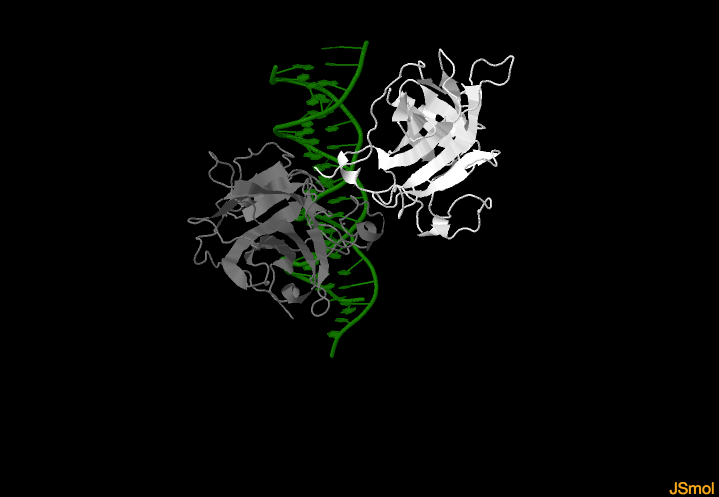
La proteína p53 presenta 4 dominios de unión a DNA (DNA binding domain o DBD) mediante los que reconoce e interactúa con secuencias específicas de DNA localizadas en las regiones promotoras de los genes diana regulados por p53. Estos genes codifican proteínas
que van a intervenir en los procesos mediados por p53. Estos cuatro dominios están asociados dos a dos formando dos dímeros (uno de estos dímeros se puede observar en la figura en la que aparece un monómero en color blanco y otro en
gris, el DNA aparece en verde).
Dos monómeros del dominio de unión a DNA de p53 interaccionando con el DNA
Las secuencias específicas que reconoce en el DNA son dos copias de la secuencia 5´-R1R2R3C4W5W6G7Y8Y9Y10-3´, donde W puede ser A o T, R puede ser A o G e Y puede ser T o C. Entre ambas secuencias puede haber un espaciador de 1 a 13 pares de bases. Hay
varias hipótesis que plantean que la presencia o ausencia del espaciador, así como la gran variedad de secuencias internas que puede adoptar la secuencia consenso, determinan un tipo de respuesta u otro por parte de p53. El hecho de
que las secuencias que reconocen los dominios de unión a DNA sean iguales reside en que esos cuatro dominios también son idénticos entre sí. Las cadenas flexibles que unen los diferentes dominios de p53 permiten que los dominios de
unión a DNA puedan unirse a diferentes variantes de esta secuencia consenso y que p53 regule la transcripción de diferentes genes a lo largo del genoma.
Estos dominios, ubicados entre los aminoácidos 94 - 294 de cada una de las cuatro cadenas polipeptídicas que forman p53, son ricos en residuos de arginina que interactúan con el DNA.
La estructura de cada dominio de unión a DNA está formada por dos láminas beta antiparalelas (en blanco en la estructura), dos grandes lazos (L2 (en azul) y L3 (en turquesa)) y un
motivo lazo-lámina-hélice (en morado) al final del dominio. La conformación de los lazos está estabilizada por un
ión zinc (en verde), coordinado tetraméricamente por tres cisteínas y una histidina (
Cys176 (en naranja),
His179) (en rosa),
Cys238 (en amarillo) y
Cys242 (en rojo)). Si tiene lugar alguna mutación en estos residuos que impida la unión del zinc, se producirá la desestabilización de todo el dominio y no será capaz de unirse al DNA.
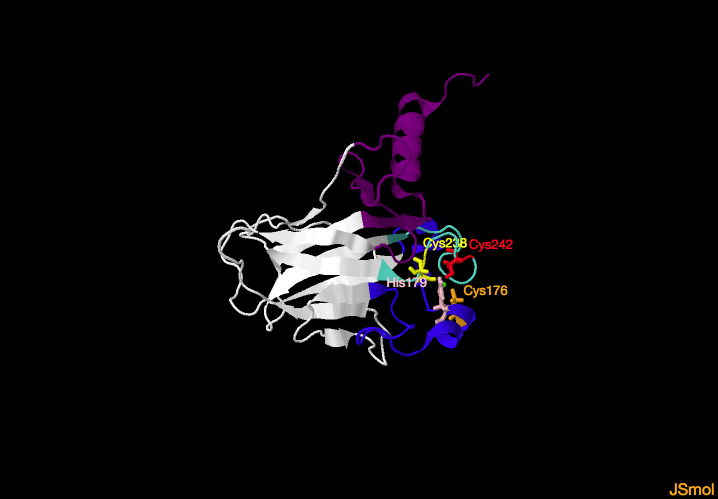
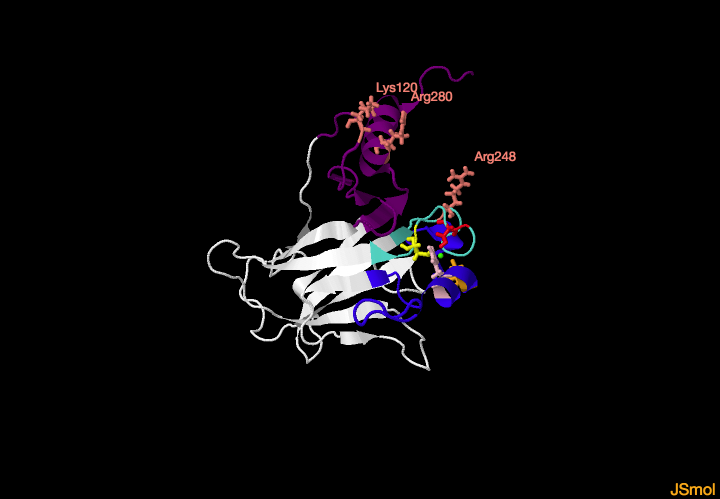
Un monómero del dominio de unión a DNA de p53
Los lazos, junto con el motivo lazo-lámina-hélice forman una superficie rica en aminoácidos básicos que se unen al DNA por dos regiones, una se une al surco mayor (por
Lys120,
Arg280) de la cadena de DNA y otra al surco menor (por
Arg248) (los tres residuos se marcan en color salmón en la estructura). Esto ocurre en cada uno de los cuatro dominios de unión a DNA que forman una proteína p53 completa. Cada dímero interacciona con 10 pares de bases de la
secuencia consenso de DNA anteriormente descrita.
Un monómero del dominio de unión a DNA de p53
MUTACIONES DE p53 Y CÁNCER
Una de las principales razones por las que el estudio de p53 adquiere gran relevancia reside en que en numerosos casos de cáncer esta proteína aparece mutada. Las mutaciones de p53 que más frecuentemente se han descrito provocan un cambio de un aminoácido
por otro diferente (mutaciones missense o de cambio de sentido) y se han localizado principalmente en el dominio de unión a DNA. El cambio de aminoácido impide el reconocimiento específico del DNA por el DBD, alterando la funcionalidad
de la proteína p53.
Podemos distinguir cuatro tipos de mutaciones según la función del aminoácido mutado:
-
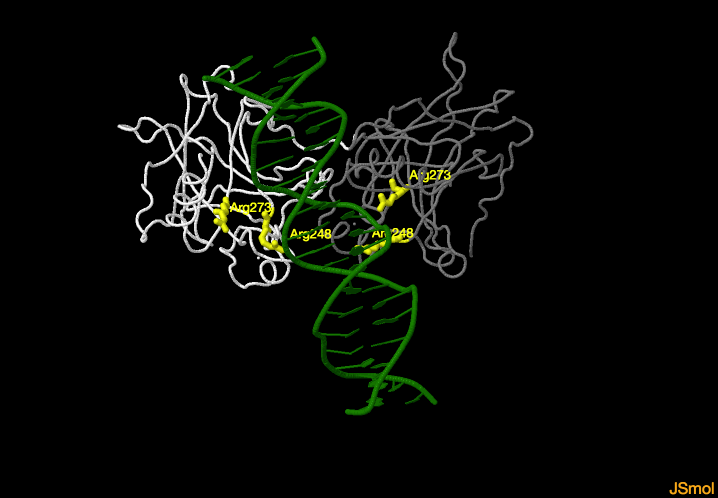
Mutaciones de aminoácidos en contacto directo con el DNA:
Arg248 (en amarillo), que interacciona con el surco menor del DNA, y
Arg273 (en amarillo).
Dímero de p53 interaccionando con el surco mayor y menor del DNA. En amarillo aminoácidos en contacto directo con el DNA.
-
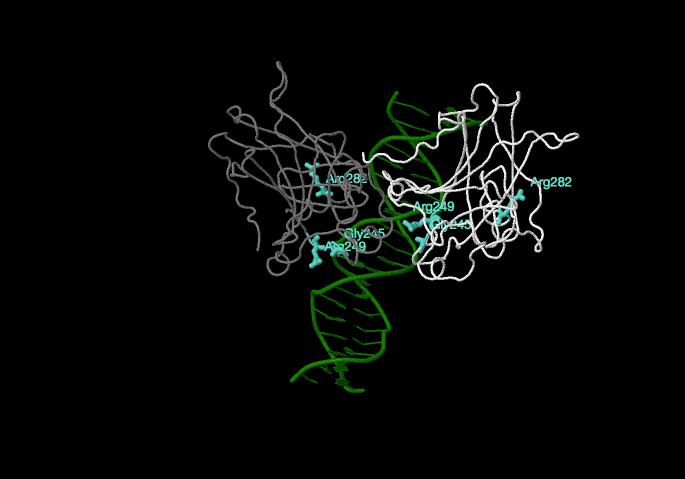
Mutaciones de aminoácidos que posicionan otros aminoácidos implicados en el contacto directo con el DNA:
Arg249, Arg282 y Gly245 (en turquesa). La mutación de estos residuos modifica la disposición de otros aminoácidos que interaccionan directamente con el DNA, llegando a alterar la estructura del dominio.
Dímero de p53 interaccionando con el DNA. En turquesa aminoácidos que posicionan otros aminoácidos implicados en el contacto directo con el DNA
-
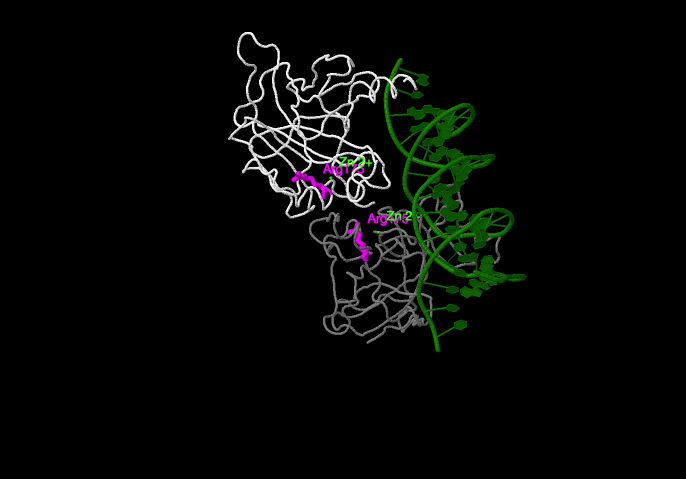
Mutaciones de aminoácidos que interaccionan con el átomo de
Zn2+ (en color lima):
Arg175 (en rosa). La mutación de este residuo desestabiliza el dominio de unión a DNA, pues su estructura se mantiene gracias a este átomo de
Zn2+ (en color lima), provocando que el DBD sea incapaz de unirse al DNA.
Dímero de p53 interaccionando con el DNA. La mutación de la Arg175, que interacciona con el Zn, desestabiliza la estructura del DBD.
-
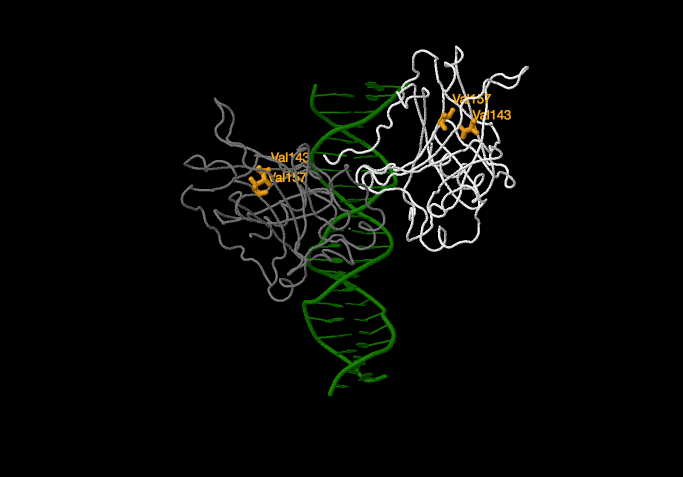
Mutaciones de aminoácidos implicados en el mantenimiento de la estructura terciaria del DBD: un ejemplo sería la
Val143 y
Val157 (en naranja). Mutaciones de estos residuos provocan un cambio estructural del dominio; en función del nuevo aminoácido insertado, este cambio estructural tendrá mayor o menor repercusión en la funcionalidad
de p53. No obstante, las mutaciones de estos aminoácidos no son tan abundantes como las de los aminoácidos mencionados anteriormente en los casos de cáncer.
Dímero de p53 interaccionando con el DNA. En naranja aminoácidos implicados en el mantenimiento de la estructura del DBD
Las mutaciones en p53 pueden originar dos efectos:
-
Pérdida de función
En situaciones normales, en una célula se expresan los dos alelos de p53 presentes. En caso de mutación en uno de ellos, se expresarán ambos igualmente, obteniéndose p53 silvestre (wtp53) y p53 mutada (mutp53).
En numerosas ocasiones, la proteína mutada ejerce una regulación dominante negativa a nivel postraduccional sobre el alelo de p53 no mutado: mutp53 puede formar un tetrámero con la wtp53, bloqueando así
la funcionalidad de la p53 silvestre en la supresión de tumores. De esta manera, una mutación en uno de los alelos puede “enmascarar” el alelo silvestre, eliminando la acción supresora de tumores al completo. Por esta razón,
las mutaciones en p53 actúan en estado heterocigoto (autosómicas dominantes) y son tan comunes en cánceres humanos.
Cabe destacar el caso del síndrome de Li-Fraumeni, que se debe a una mutación missense en p53 de células germinales, de forma que un alelo queda inactivado en todas las células del organismo. Esto hace
que los individuos que padecen este síndrome tengan una mayor probabilidad de desarrollar un gran número de cánceres: sarcomas, tumores cerebrales y leucemias, entre otros.
Para conocer más información acerca de esta patología, puede resultar de interés la consulta del siguiente enlace:
Artículo (Síndrome de Li-Fraumeni)
-
Ganancia de función
Mutp53, también gana una función protooncogénica a través de distintas acciones sobre genes diana diferentes a los de la wtp53. Mutp53 puede interaccionar con un gran número de factores de transcripción
y cofactores, incrementando o disminuyendo su acción transcripcional para promover la progresión del tumor.
REGULACION
La proteína p53 se encuentra regulada principalmente por
MDM2, una ubiquitina E3 ligasa que se encarga de conducir la wtp53 a su degradación en el proteasoma en condiciones normales (manteniendo niveles bajos de ésta en los tejidos), a través de la interacción con determinados aminoácidos
del
dominio de transactivación de p53. A su vez, MDM2 es codificada por genes controlados por p53, de manera que se establece un bucle de retroalimentación negativa entre ambos. Cuando p53 se encuentra mutado, MDM2 presenta menor
capacidad de interacción con p53 para su degradación y, además, mutp53 no puede inducir su transcripción. De esta forma, mutp53 aumenta su concentración y se acumula en los tejidos, pudiendo desencadenar en los fenómenos de ganancia
de función mencionados previamente.
MDM2 interaccionando con la parte del dominio de transactivación encargada de su unión
No obstante, también juega un papel fundamental MDMX, un homólogo de MDM2 que regula negativamente p53 a través de la modulación de su actividad como factor de transcripción.
Para conocer más en profundidad estos dos reguladores y otras E3 ligasas puede ser útil la consulta del siguiente enlace.
Artículo (MDM2-MDMX/P53)
Guion elaborado por Víctor Álvaro Sánchez, Aitana Avendaño Pomares, Ana Carbajo Uña, Andrea Fuentes Terrón y Diego Rodríguez Manzano, alumnos de Química e Ingeniería de Proteínas, 3er curso del Grado en Biotecnología, curso 2017/18.
Referencias
Melero del Río, R. Estudio de la estructura cuaternaria de p53 y el complejo p53/ADN mediante microscopía electrónica. (2010).
Wang, X. & Jiang, X. Mdm2 and MdmX partner to regulate p53. FEBS Letters 586, 1390–1396 (2012).
Yue, X. et al. Mutant p53 in Cancer: Accumulation, Gain-of-Function, and Therapy. Journal of Molecular Biology 429, 1595–1606 (2017).
López, M., Anzola, M., Cuevas-Salazar, N., Aguirre, J. M. & de Pancorbo, M. M. p53, un gen supresor tumoral. Gaceta médica de Bilbao 98, 21–27 (2001).
Chompret, A. The Li–Fraumeni syndrome. Biochimie 84, 75–82 (2002).