Introducción
Las inteínas son un ejemplo de cómo las células producen varias proteínas a partir de un único gen. Concretamente, las inteínas gracias a su actividad autoproteolítica catalizan la escisión de una secuencia de proteína intermedia
(la propia inteína) de un precursor proteico, y la unión posterior de los fragmentos de proteína flanqueantes (exteínas) (Figura 1). Este sistema se asemeja considerablemente al splicing del RNA, solo que
se trata de proteínas en vez de ácido nucleicos, y se conoce como autosplicing o autoproteolisis. El resultado es la formación de un enlace peptídico entre la N-exteína y la C-exteína, dando lugar a la proteína madura,
y una inteína libre. El splicing proteico mediado por inteínas es espontáneo; no requiere factor externo ni fuente de energía, solo el plegamiento del dominio inteína.
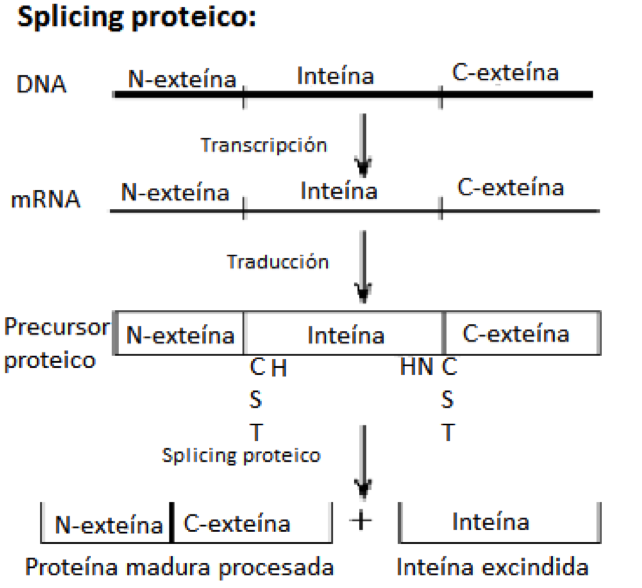 Figura 1: Esquema del empalme o splicing proteico. Se muestran los residuos conservados de los extremos de la inteína: Cys, Ser o Thr e His, en el extremo N-terminal; His, Asn y Cys o Ser o Thr en extremo C-terminal.
Figura 1: Esquema del empalme o splicing proteico. Se muestran los residuos conservados de los extremos de la inteína: Cys, Ser o Thr e His, en el extremo N-terminal; His, Asn y Cys o Ser o Thr en extremo C-terminal.
Las inteínas se han llamado a veces “intrones proteicos”. Es conveniente destacar que, en este proceso, la secuencia intermedia (inteína) está presente en el mRNA maduro y se traduce para formar la proteína precursora.
Mecanismo
El mecanismo de autoempalme de proteínas implica una serie de reacciones que dan como resultado la rotura de dos enlaces peptídicos en las uniones inteína-exteína y la formación de un nuevo enlace peptídico entre las N- y C-exteínas.
Este proceso se inicia mediante la activación del enlace peptídico que une la N-exteína y el extremo N-terminal de la inteína. Prácticamente todas las inteínas tienen una
cisteína o serina en su extremo N-terminal (Bloque A) (Figura 2) que ataca al carbono carbonílico del residuo C-terminal de la N-exteína. Este proceso se ve facilitado por una treonina e histidina conservadas en el Bloque B
(o motivo
TXXH), junto con un
aspartato encontrado comúnmente en el Bloque F, y da como resultado la formación de un intermediario (tio)éster lineal.
 Figura 2: Esquema de la localización de aminoácidos implicados en el mecanismo de acción de la inteína.
Figura 2: Esquema de la localización de aminoácidos implicados en el mecanismo de acción de la inteína.
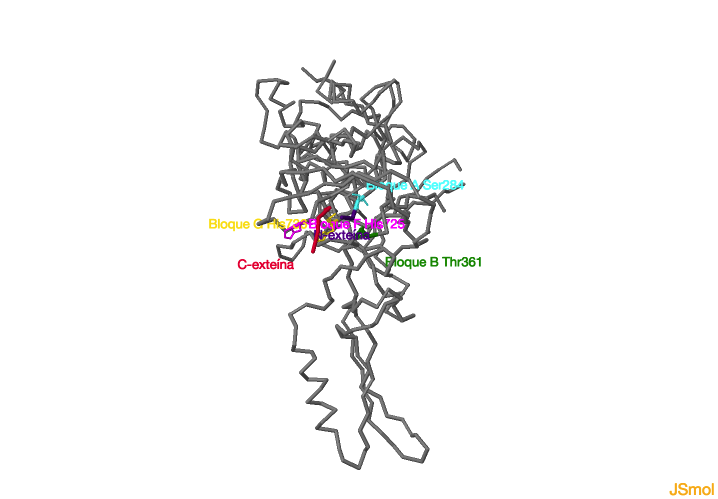
En el siguiente enlace se muestran los aminoácidos de la inteína pertenecientes a los distintos bloques implicados en la catálisis: bloque
A-Ser284 (cian),
bloque B-Thr361 (verde),
bloque F-His725 (rosa),
bloque G-His736 (amarillo). También se marcan dos residuos de la
N-exteína (morado) y cuatro residuos de la
C-exteína (rojo).
Aminoácidos de la inteína implicados en la reacción de autosplicing.
En la primera reacción del mecanismo de acción, la cisteína o serina del extremo N-terminal de la
inteína ataca al carbono carbonílico del residuo C-terminal de la
N-exteína (Figura 3), formándose un enlace tioéster entre ambos residuos.
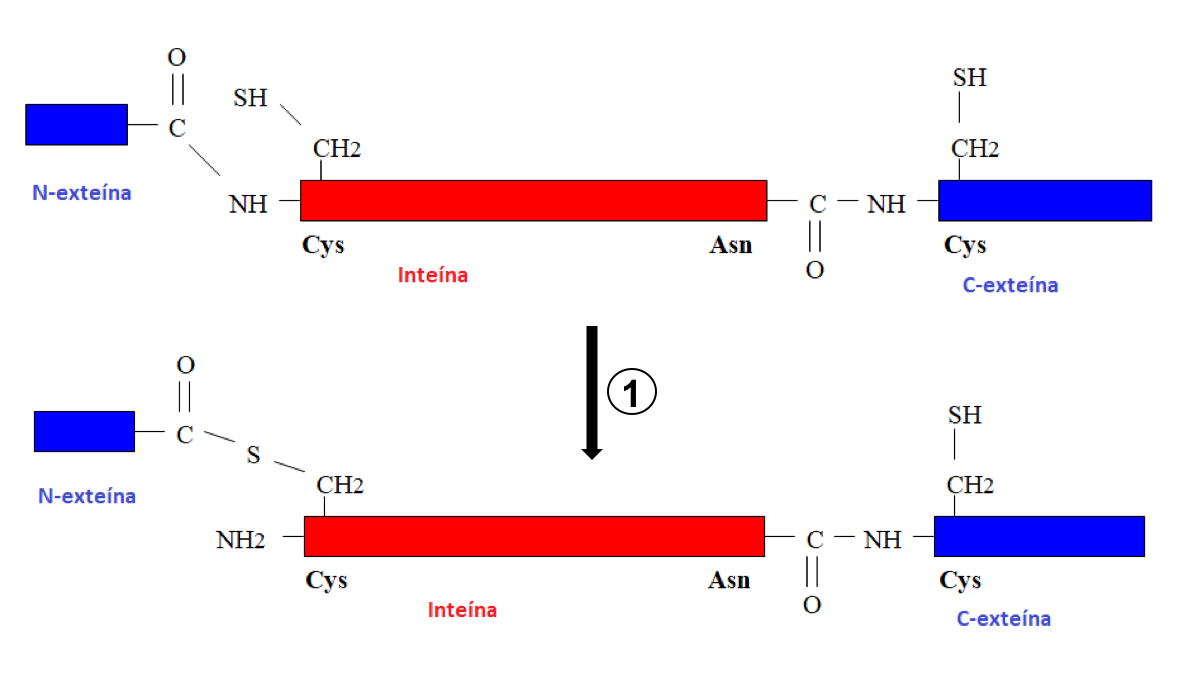 Figura 3: Primer paso del mecanismo de autosplicing: formación de un intermediario tioéster.
Figura 3: Primer paso del mecanismo de autosplicing: formación de un intermediario tioéster.
A continuación, este intermediario se ve sujeto a una trans-(tio)esterificación por ataque nucleofílico del primer residuo de C-exteína (denominado +1), que invariablemente es una cisteína, serina o treonina (Figura 1).
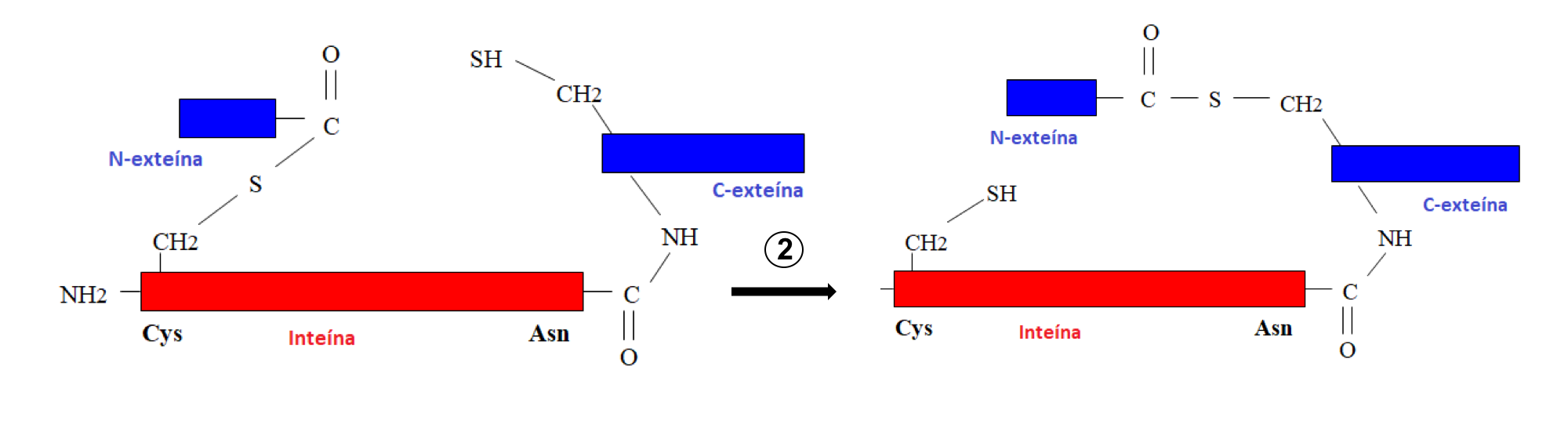 Figura 4: Segundo paso del mecanismo de autosplicing: transesterificación.
Figura 4: Segundo paso del mecanismo de autosplicing: transesterificación.
El intermediario (tio)éster ramificado resultante se resuelve en la tercera reacción mediante una transformación única: la formación de un enlace succinimida que determina la ciclación de la
asparagina C-terminal altamente conservada de la inteína (
Bloque G en la Figura 2). Este proceso se ve facilitado por la
histidina del bloque F, que se encuentra en un motivo HNF también altamente conservado, y la penúltima
histidina del bloque G; también puede implicar al
aspartato del bloque Fz. Esta reacción de formación de succinimida escinde la inteína del complejo reactivo y deja atrás las exteínas unidas mediante un enlace no peptídico.
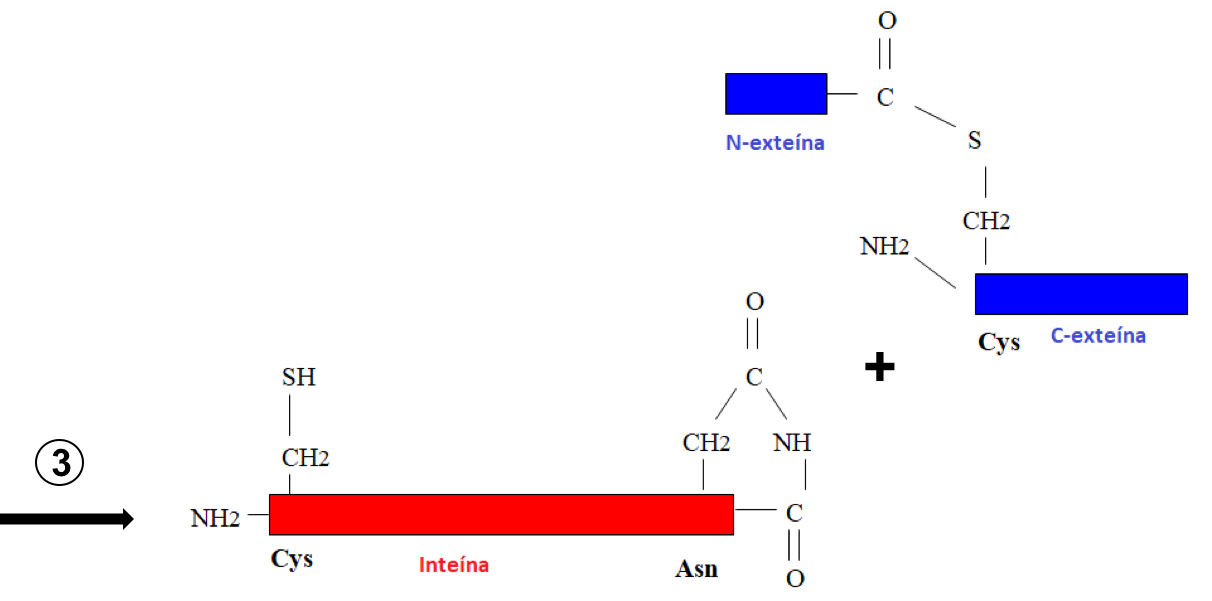 Figura 5: Tercer paso del mecanismo de autosplicing: ciclación de Asn y excisión de exteínas.
Figura 5: Tercer paso del mecanismo de autosplicing: ciclación de Asn y excisión de exteínas.
Esta estructura se reorganiza rápidamente de manera independiente de la inteína formándose un enlace peptídico estable entre las N- y C-exteínas (Figura 6). Además, la succinimida en el C-terminal de la inteína escindida se
hidrolizará lentamente.
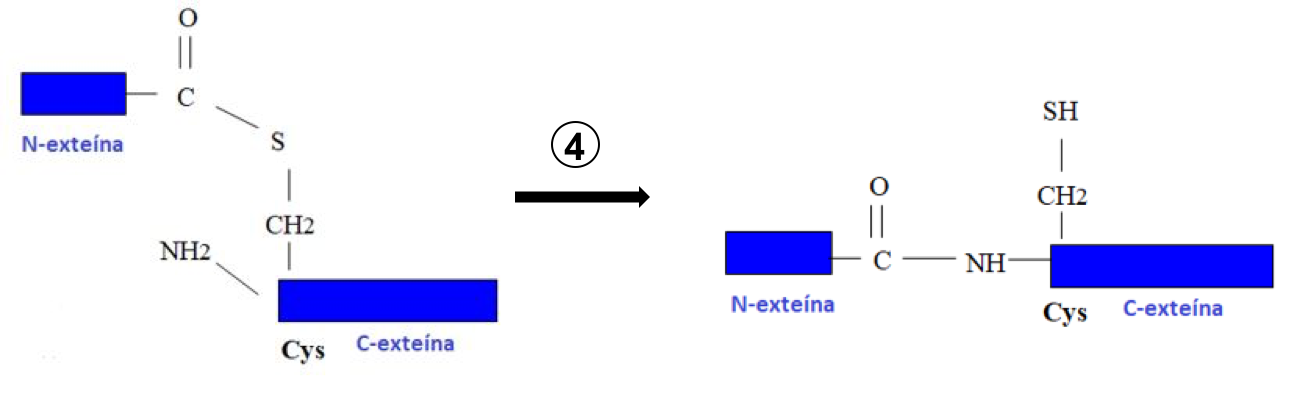 Figura 6:Cuarto paso del mecanismo de autosplicing: formación del enlace peptídico entre las dos exteínas.
Figura 6:Cuarto paso del mecanismo de autosplicing: formación del enlace peptídico entre las dos exteínas.
En la siguiente animación se resume el mecanismo de autoproteolisis catalizado por las inteínas.
Mecanismo de autosplicing proteico (vídeo). (Tomado de Southworth MW, en InBase).
Estructura
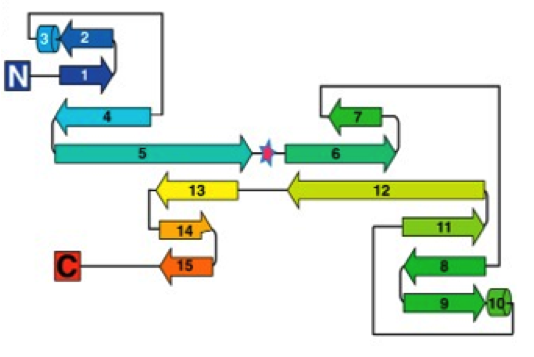
Mediante RMN y cristalografía de rascrolling="no"s X se ha observado que las inteínas presentan un plegamiento conservado en forma de herradura. El plegamiento consta principalmente de hojas β, bucles y dos hélices cortas, y tiene
pseudo doble simetría (el eje de pseudosimetría se marca con una estrella en la Figura 7).
Figura 7: Estructura secundaria de la inteína (tomado de Eryilmaz et al., 2014).
El plegamiento de inteínas tiene dos características notables:
- - Ambos extremos de la inteína, unidos a las dos exteínas, se localizan muy próximos (
<10 A) para facilitar el empalme.
- - Alrededor de estos extremos se encuentran múltiples sitios activos similares a los de proteasas que llevan a cabo cada uno de los pasos químicos implicados en el splicing de proteínas.
El plegamiento de una inteína se acopla a las reacciones catalizadas: la estructura inicial facilita la primera etapa de splicing, y después de cada etapa se provocan cambios conformacionales locales, que afectan al plegamiento
del centro catalítico. En definitiva, el plegamiento de la inteína está acoplado con la reacción de autosplicing.
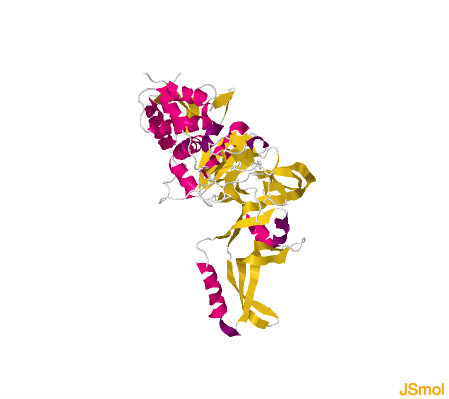
Comparando las estructuras mostradas en los siguientes enlaces, podemos ver cómo cambia la estructura de la
inteína (gris) y de las exteínas (
rojo y
azul) tras el proceso de splicing.
A)
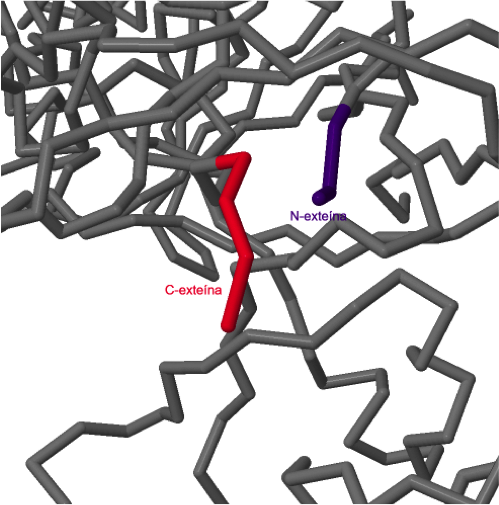
B)
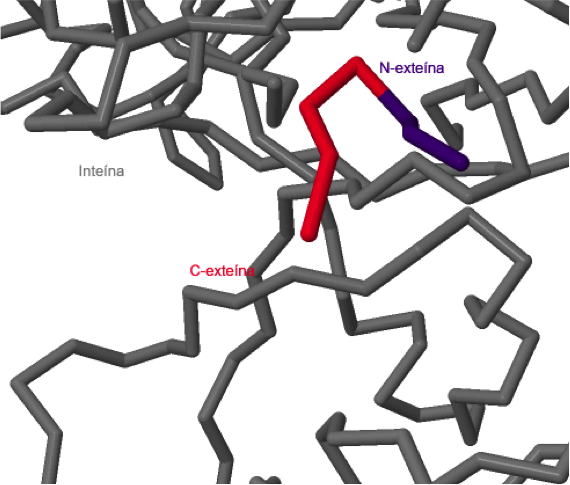
Estructura de la inteína y exteínas, A) previa al proceso de splicing, B) estructura de la inteína y exteínas después del proceso de splicing.
Inteínas con actividad endonucleasa Homing
Muchas inteínas tienen insertado dentro de su dominio de autoprocesamiento otro módulo funcional llamado dominio de endonucleasa homing (HED). Los HED hacen rupturas de DNA bicatenario en secuencias de reconocimiento específicas codificadas
dentro de alelos libres de inteína, localizadas en genes homólogos a la secuencia de la exteína pero sin ella. Estas roturas inician un mecanismo de reparación normal en el que se utiliza el gen que contiene la secuencia de inteína,
lo que da como resultado la integración de dicho gen en un genoma libre de ellos. Esto no supone un problema, ya que la inteína se separará de la proteína cuando se produzca el autosplicing proteico (Figura 7).
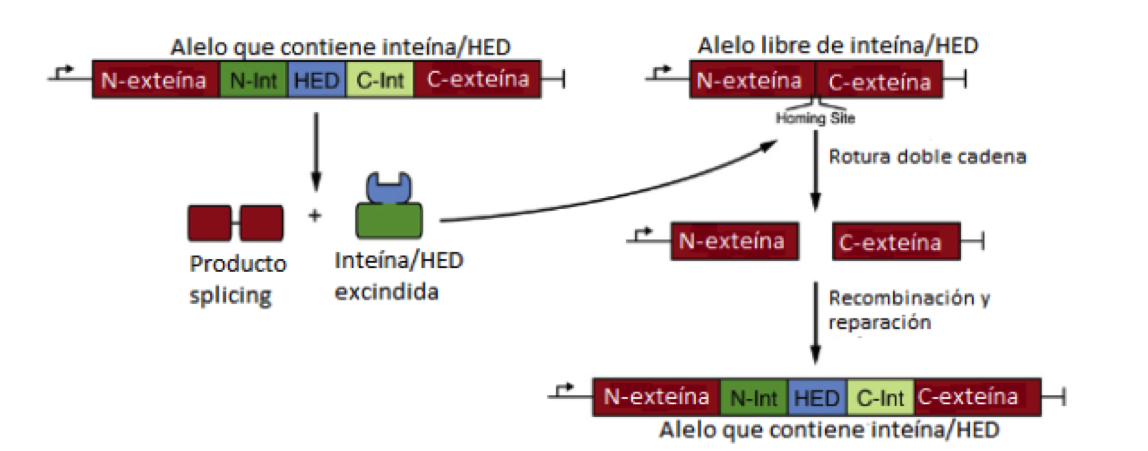 Figura 8: Inteínas con actividad endonucleasa Homing (tomado de Shah & Muir, 2014).
Figura 8: Inteínas con actividad endonucleasa Homing (tomado de Shah & Muir, 2014).
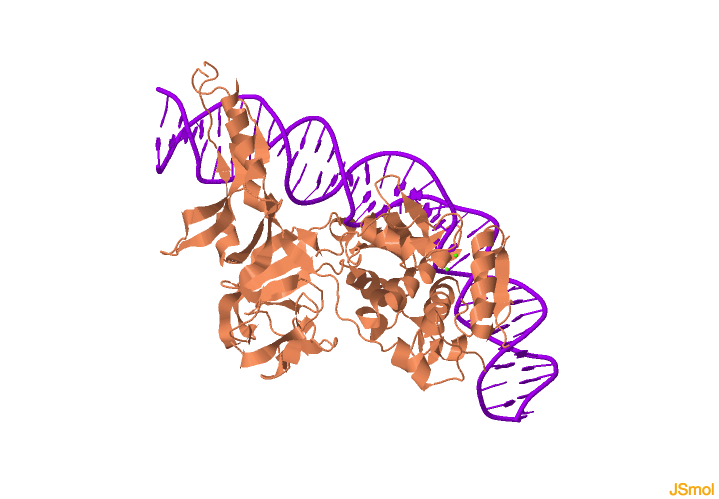
En el siguiente enlace se observa la estructura de la inteína unida al DNA:
Estructura de la inteína unida al DNA.
Aplicaciones
Como acabamos de ver, las inteínas catalizan la rotura y formación de enlaces peptídicos. Esta propiedad permite que inteínas aisladas puedan introducirse o empalmarse en proteínas extrañas heterólogas si se colocan en un entorno
de proteínas huésped compatible. En definitiva, las inteínas son herramientas importantes en el campo de la química e ingeniería de proteínas que pueden usarse para la investigación básica y con fines terapéuticos, aunque son
un campo que aún está por explorar.
Ejemplos de lo que puede conseguirse mediante estas proteínas modificadas genéticamente son:
- - Purificación de proteínas sin etiqueta.
- - Semi-síntesis de proteínas in vitro e in vivo.
- - Ciclación de proteínas y péptidos.
- - “Empalme proteico condicionado” (conditional protein splicing o CPS), es decir, activación o inhibición del splicing por un modulador externo.
Guion elaborado por Gema de Miguel Muguerza, Pablo Olivas del Saz, Laura Prieto López y Mónica Yagüe Sancho, alumnos de Química e Ingeniería de Proteínas, 3er curso del Grado en Biotecnología, curso 2017/18.
Cuestiones
Responder al cuestionario correspondiente en la página de Studium
Referencias
Anraku, Y. & Satow, Y. (2009) Protein-splicing reaction via a thiazolidine intermediate: crystal structure of the VMA1-derived endonuclease bearing the N and C-terminal propeptides. J Mol Biol. 316(4):919-29. https://www.ncbi.nlm.nih.gov/pmc/articles/PMC3621562/figure/f2-pjab-85-409/
Anraku, Y. & Satow, Y. (2009). Reflections on protein splicing: structures, functions and mechanisms. Proceedings of the Japan Academy. Series B, Physical and Biological Sciences 85(9), 409–421. http://doi.org/10.2183/pjab.85.409
Eryilmaz, E., Shah, N. H., Muir, T. W. & Cowburn, D. (2014). Structural and Dynamical Features of Inteins and Implications on Protein Splicing. The Journal of Biological Chemistry 289(21), 14506–14511. http://doi.org/10.1074/jbc.R113.540302
Goodsell D., RCSB PDB Molecule of the Month doi:10.2210/rcsb_pdb/mom_2010_11
InBase, The Intein Database and Registry, http://www.inteins.com/
PDB www.rcsb.org, H.M. Berman, J. Westbrook, Z. Feng, G. Gilliland, T.N. Bhat,
H. Weissig, I.N. Shindyalov, P.E. Bourne (2000) The Protein Data Bank URL: Nucleic Acids Research, 28:235-242.
Shah, N.H. & Muir, T.W. (2014). Inteins: nature's gift to protein chemists. Chem. Sci. 5, 446- 461